- 고객지원
- 업무안내│제품문의
- 전화번호│02-2081-2510
- Email │support@takara.co.kr
- 대전지사
- 업무안내│대전/충청지역 주문, 제품문의
- 전화번호│042-828-6525
- Email │tkbd@takara.co.kr
- 업무시간안내
- [ 평 일 ] 09 : 00 ~ 18 : 00 │ [ 점심시간 ] 12 : 00 ~ 13 : 00
- 토·일요일, 공휴일은 휴무입니다.
[Cancer Research] Cancer Genomics & Epigenomics

Using Unique Molecular Tags (UMTs) in NGS experiments
Next-generation sequencing (NGS) 기술은 빠르게 발전하고 있고, 저변이 대립유전자 (low-frequency alleles)를 확실하게 분석하거나 분자 수준을 구분해내는 능력은 매우 민감한 NGS 기반 분석법을 개발하는데 중요하다. Unique Molecular Tags (UMTs)는 error correction과 molecular de-duplication를 제공함으로써 매우 유용하게 사용될 수 있다.
NGS를 진행함에 있어 PCR, sequencing, base calling 등의 과정에서 error가 종종 발생될 수 있다. UMTs를 추가하여 error를 제거하고, 위양성의 백그라운드를 낮출 수 있어, 높은 민감도와 특이성으로 낮은 빈도의 대립유전자도 신뢰도 있게 검출해 낼 수 있다. 게다가 실험 초기 단계에서 DNA 각각에 UMT를 라벨링 함으로써 분석 과정에서 PCR duplicate를 제거하거나 molecular duplicates를 구별해낼 수 있어, 결과적으로 사용된 샘플의 초기 분자 수를 계산할 수 있다.
What are UMTs?
UMTs는 증폭 전 각각의 DNA fragment를 tag하는 unique sequence로, library 제작 과정이나 target enrichment, 데이터 분석 과정에서의 fragment를 추적할 수 있게 한다. ThruPLEX® Tag-seq Kit 내에는 각각의 fragment를 서로 다른 서열로 labeling하기에 충분한 1,600만개 이상의 UMT가 제공된다.
How do UMTs work?
다카라바이오는 ThruPLEX® Tag-seq Kit 내에 포함되어 있는 독자적인 기술의 stem-loop adapter가 degenerate base로 구성된 unique sequence를 포함함으로써 tag 역할을 할 수 있도록 설계하였다. 이렇게 tagging된 adapter는 library 제작 과정의 ligation 스텝에서 부착되게 되고, 각각의 DNA를 labeling한다. 그 후, UMT를 기반으로 한 sequencing reads를 그룹화함으로써, 데이터 처리 과정에서 증폭 산물을 분별해낼 수 있다.

그림 1. UMT를 이용한 DNA의 그룹화
The second and third tag sequences represented by the teal, red, cyan, green, pink, and yellow sections (above) are the inserted markers; by comparing the markers, the molecules can be identified as either identical or unique. In the example above, the five individual colors on top indicate five unique DNA molecules, while the five below, all of which include an identical yellow tag, are determined to be duplicates.
데이터 분석 과정을 통해 분류된 각 증폭 read들을 비교하고, PCR 산물이나 sequencing 오류를 제거함으로써 공통되는 서열을 얻어낼 수 있다.
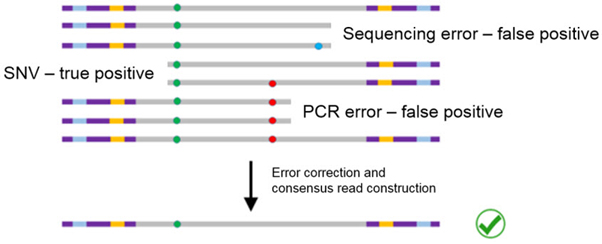
그림 2. Tag를 이용한 error와 위양성 서열 검출
By comparing the different sequencing results and eliminating the error strands, a consensus read can be determined.
When should I use UMTs?
일반적인 NGS library를 이용하면 약 5% 정도의 대립 유전자 변이를 검출하기에 충분하나, 대부분의 PCR이나 sequencing 과정의 error 또한 이 이하 수준으로 발생한다. 1%나 그 이하로 발생하는 낮은 빈도의 대립 유전자 돌연변이를 분석하고자 하는 연구자들은 tagging을 포함하는 library를 이용함으로써 5% 이하로 발생하는 돌연변이를 확인할 수 있어 매우 유용하다. 다만, 더 높은 특이성과 민감도를 분석하기 위해서는 보다 많은 분석 비용이 요구된다.
What else do I need to consider when sequencing libraries with UMTs?
UMT의 장점은 redundant sequencing이나 deep sequencing을 진행할 때 가장 큰 힘을 발휘한다. 분석하고자 하는 각각의 고유 DNA fragment는 여러 차례 sequencing과 duplicate reads를 비교하는 과정을 통해 PCR과 sequencing error를 제거함으로써 기존의 고유 서열을 알아낼 수 있다. 이와 같은 분석을 위해서는 특정한 데이터 분석 툴이 필요하며, 다카라바이오는 이를 위한 Cogent NGS Immune Profiler Software를 무상으로 제공하고 있다. 다만, sequencing 과정에서 데이터 분석 양이 너무 많아질 수 있기에, 각각의 샘플에서 확인되는 sequencing reads를 낮추기 위해서 targeted sequencing을 진행하는 것이 일반적이다.
[원문] Using Unique Molecular Tags (UMTs) in NGS experiments
Next-generation sequencing (NGS) 기술은 빠르게 발전하고 있고, 저변이 대립유전자 (low-frequency alleles)를 확실하게 분석하거나 분자 수준을 구분해내는 능력은 매우 민감한 NGS 기반 분석법을 개발하는데 중요하다. Unique Molecular Tags (UMTs)는 error correction과 molecular de-duplication를 제공함으로써 매우 유용하게 사용될 수 있다.
NGS를 진행함에 있어 PCR, sequencing, base calling 등의 과정에서 error가 종종 발생될 수 있다. UMTs를 추가하여 error를 제거하고, 위양성의 백그라운드를 낮출 수 있어, 높은 민감도와 특이성으로 낮은 빈도의 대립유전자도 신뢰도 있게 검출해 낼 수 있다. 게다가 실험 초기 단계에서 DNA 각각에 UMT를 라벨링 함으로써 분석 과정에서 PCR duplicate를 제거하거나 molecular duplicates를 구별해낼 수 있어, 결과적으로 사용된 샘플의 초기 분자 수를 계산할 수 있다.
What are UMTs?
UMTs는 증폭 전 각각의 DNA fragment를 tag하는 unique sequence로, library 제작 과정이나 target enrichment, 데이터 분석 과정에서의 fragment를 추적할 수 있게 한다. ThruPLEX® Tag-seq Kit 내에는 각각의 fragment를 서로 다른 서열로 labeling하기에 충분한 1,600만개 이상의 UMT가 제공된다.
How do UMTs work?
다카라바이오는 ThruPLEX® Tag-seq Kit 내에 포함되어 있는 독자적인 기술의 stem-loop adapter가 degenerate base로 구성된 unique sequence를 포함함으로써 tag 역할을 할 수 있도록 설계하였다. 이렇게 tagging된 adapter는 library 제작 과정의 ligation 스텝에서 부착되게 되고, 각각의 DNA를 labeling한다. 그 후, UMT를 기반으로 한 sequencing reads를 그룹화함으로써, 데이터 처리 과정에서 증폭 산물을 분별해낼 수 있다.

그림 1. UMT를 이용한 DNA의 그룹화
The second and third tag sequences represented by the teal, red, cyan, green, pink, and yellow sections (above) are the inserted markers; by comparing the markers, the molecules can be identified as either identical or unique. In the example above, the five individual colors on top indicate five unique DNA molecules, while the five below, all of which include an identical yellow tag, are determined to be duplicates.
데이터 분석 과정을 통해 분류된 각 증폭 read들을 비교하고, PCR 산물이나 sequencing 오류를 제거함으로써 공통되는 서열을 얻어낼 수 있다.

그림 2. Tag를 이용한 error와 위양성 서열 검출
By comparing the different sequencing results and eliminating the error strands, a consensus read can be determined.
When should I use UMTs?
일반적인 NGS library를 이용하면 약 5% 정도의 대립 유전자 변이를 검출하기에 충분하나, 대부분의 PCR이나 sequencing 과정의 error 또한 이 이하 수준으로 발생한다. 1%나 그 이하로 발생하는 낮은 빈도의 대립 유전자 돌연변이를 분석하고자 하는 연구자들은 tagging을 포함하는 library를 이용함으로써 5% 이하로 발생하는 돌연변이를 확인할 수 있어 매우 유용하다. 다만, 더 높은 특이성과 민감도를 분석하기 위해서는 보다 많은 분석 비용이 요구된다.
What else do I need to consider when sequencing libraries with UMTs?
UMT의 장점은 redundant sequencing이나 deep sequencing을 진행할 때 가장 큰 힘을 발휘한다. 분석하고자 하는 각각의 고유 DNA fragment는 여러 차례 sequencing과 duplicate reads를 비교하는 과정을 통해 PCR과 sequencing error를 제거함으로써 기존의 고유 서열을 알아낼 수 있다. 이와 같은 분석을 위해서는 특정한 데이터 분석 툴이 필요하며, 다카라바이오는 이를 위한 Cogent NGS Immune Profiler Software를 무상으로 제공하고 있다. 다만, sequencing 과정에서 데이터 분석 양이 너무 많아질 수 있기에, 각각의 샘플에서 확인되는 sequencing reads를 낮추기 위해서 targeted sequencing을 진행하는 것이 일반적이다.
Code |
제품명 |
용량 |
R400584 |
24회 |
|
R400742 |
24회 |
[원문] Using Unique Molecular Tags (UMTs) in NGS experiments