- 고객지원
- 업무안내│제품문의
- 전화번호│02-2081-2510
- Email │support@takara.co.kr
- 업무시간안내
- [ 평 일 ] 09 : 00 ~ 18 : 00 │ [ 점심시간 ] 12 : 00 ~ 13 : 00
- 토·일요일, 공휴일은 휴무입니다.
[Overview] In-Fusion® Cloning

[Overview] In-Fusion® Cloning
1. In-Fusion® cloning 기술 개요
Ligation-independent cloning (LIC) 방법 중의 하나로써, 3’ → 5’ exonuclease 활성을 가지는 In-Fusion® 효소를 이용해 DNA 단편 간의 상동서열 (약 15 bp)를 융합시켜 cloning하는 기술로, ligase 없이도 쉽고 정확하게 seamless한 산물을 얻을 수 있다.
2. In-Fusion® cloning 특징
반응 시간 |
|
효율 |
|
활용성 |
|
3. In-Fusion® cloning 실험 과정

4. In-Fusion® cloning primer design
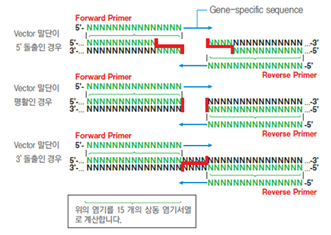
PCR primer는 gene specific sequence*와 vector 상동서열을 포함하도록 제작하며, 각 vector 말단의 형상에 따라 primer를 디자인한다.
* Gene specific sequence의 자세한 조건은 매뉴얼을 참조하시오.
* In-Fusion® primer 디자인 툴 (클릭)
5. In-Fusion® cloning 제품 선택가이드
특징 |
||
형태 |
액상 |
동결 건조 |
Control vector, insert 포함 |
O |
O |
원하는 reaction volume 혹은 plate/tube 등에서 사용 |
O |
|
HTP 적용을 위한 편리한 scale up |
O |
O |
1회분으로 분주 및 동결 건조되어, handling error 최소화 |
|
O |
실온에서 보관 가능 |
|
O |
6. 타 PCR cloning 방식과의 비교 (vs. Gibson assembly)
|
In-Fusion® Snap Assembly |
Gibson assembly-based cloning |
Colony 수 |
다소 복잡한 실험 (예 - multiple cloning, large insert/vector 등)에서도 타 방식에 비해 colony가 많이 형성됨 |
복잡한 실험의 경우 colony가 적게 형성되거나, 형성되지 않음 |
Nick 수복 과정 |
Polymerase를 이용하지 않고, competent cell에서 nick을 수복하여, 이로 인한 불필요한 염기 추가 등의 에러 최소화 |
DNA polymerase가 enzyme mix 내 포함되어 있어, error가 수복 부위에서 발생할 수 있음. |
Ligase를 사용하지 않아, self ligation 등의 백그라운드가 나타날 확률이 적음 |
in vitro ligation을 이용하므로, empty vector 등 백그라운드가 나타날 확률이 높음. |
|
반응 시간 |
15 분 |
~ 60 분 |
A-overhang insert 교정 |
3’ → 5’ exonuclease 활성을 이용해, PCR 과정에서 임의로 부가된 A 1염기의 교정이 가능함 |
5’ → 3’ exonuclease 활성을 이용하기에, PCR 과정에서 임의로 부가된 A 1염기에 대한 교정이 불가능해, 정확도가 떨어짐 |
PCR primer 디자인 |
15 bp overlap으로, PCR 조건 설정 시 유리 |
일반적으로 20 ~ 30 bp overlap |
7. 참고 자료
- So Easy Product, So Easy Cloning (클릭)
- In-Fusion® Cloning - Applications Collections (클릭)
- [FAQ] In-Fusion® Cloning (클릭)
- [Tips] In-Fusion® Cloning (클릭)
- In-Fusion® Cloning - 실제 사용 후기 1탄 (클릭)
- In-Fusion® Cloning - 실제 사용 후기 2탄 (클릭)