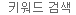- 고객지원
- 업무안내│제품문의
- 전화번호│02-2081-2510
- Email │support@takara.co.kr
- 업무시간안내
- [ 평 일 ] 09 : 00 ~ 18 : 00 │ [ 점심시간 ] 12 : 00 ~ 13 : 00
- 토·일요일, 공휴일은 휴무입니다.
Home >
전제품보기 >
기기 · 소모품 >
NGS 관련장비 >
SMARTer ICELL8 single cell system으로 full-length single cell RNA-seq
SMARTer ICELL8 single cell system으로 full-length single cell RNA-seq

본 데이터는 SMARTer ICELL8 Single-Cell System을 사용하여 수백 개의 single cell에서 full-length sequence 정보를 얻을 수 있는 고품질의 RNA-seq library를 생성할 수 있음을 보여줍니다. SMARTer ICELL8 system을 사용한 full-length single cell RNA-seq에 대한 방법은 cDNA 합성 및 PCR 증폭을 포함하여 SMARTer ICELL8 chip에서 Illumina NGS library 준비에 필요한 모든 반응을 수행할 수 있게 하며, chip으로부터의 결과물은 sequencing이 가능한 library이다. SMART template-switching 기술을 결합하여 고품질의 cDNA 생산을 보장하는 동시에 다양한 single cell 분석을 높은 감도 및 높은 재현성으로 수행할 수 있습니다.
Full-length RNA-seq library preparation workflow
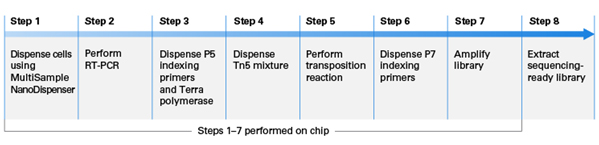
그림1. SMARTer ICELL8 library preparation workflow for full-length single-cell RNA-seq.
살아있는 single cell이 포함된 well만을 선택한 후, cDNA는 one-Step RT-PCR 반응(Step 2)으로 oligo-dT priming을 통해 합성됩니다. 이어지는 library 제작을 위해 ICELL8 chip의 72개 행 각각은 다른 index를 갖고있는 모든 well에 P5 index primers로 index가 분주되고, Terra polymerase와 reaction buffer(Step 3)가 분주됩니다. Transposition enzyme 및 reaction buffer(Tn5 mixture)를 선택된 well에 분주하고(Step 4), transposition reaction을 수행한다(Step 5). P7 index primer는 well에 분배되고, chip상의 72개 열 각각은 상이한 index를 받게된다(Step 6). Illumina sequencer에서 sequencing이 가능하도록 만들어진 library는 amplification(7 Step)되고 chip에서 회수하면서 pooling됩니다. pooled library는 Illumina sequencer에 load하기전 수차례 bead 정제와 정량을 마친 후 sequencing 합니다.
Gene-body coverage
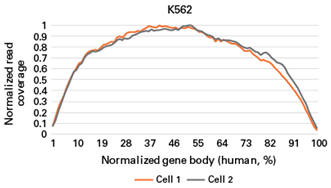
그림 2. Assessment of gene-body coverage for representative samples.
Human genome에 의해 코딩된 유전자 구조의 길이가 표준화되었고, 상이한 2개의 분리된 K562 cell로부터 mapping된 판독 범위가 표준화 된 유전자 본체를 따라 플로팅되었다. (표준화된 유전자 본체 내의 위치는 X축상의 백분율로 표시된다). corverage (Y축에서 0 대 1의 척도로 표현됨)는 정규화된 gene body를 따라 관찰된 최대 corverage와 관련하여 정규화되었다.
Read-mapping distributions for data generated from single cells
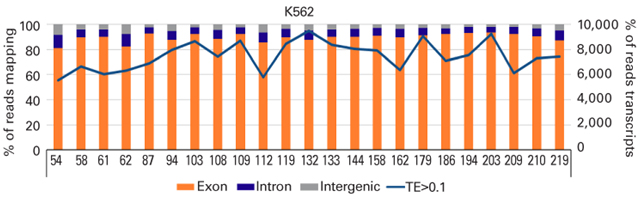
그림 3. Sequencing metrics for selected samples (K562 cells).
Human genome의 표시된 영역에 mapping되는 read의 백분율을 나타내는 막대 그래프(Exon(주황색), Intron(파란색) 또는 intergenic region(회색)) histogram에 겹쳐진 진한 파란색 선은 각 well에 대해 RPKM value >0.1로 확인된 transcript의 수를 나타냅니다. 각 histogram의 하단에 있는 숫자는 데이터를 얻은 single cell이 위치한 chip상의 well의 위치를 나타냅니다.
Relationship between read depth and number of transcripts identified
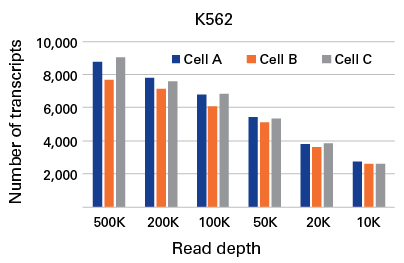
그림 4. Relationship between read depth and number of transcripts identified for three single-cell containing wells.
상기의 그래프는 3개의 K562 cell이 들어있는 well 각각에 대한 다양한 read depths에서 확인된 transcript의 수를 나타낸다.
Reproducibility of gene expression measurements
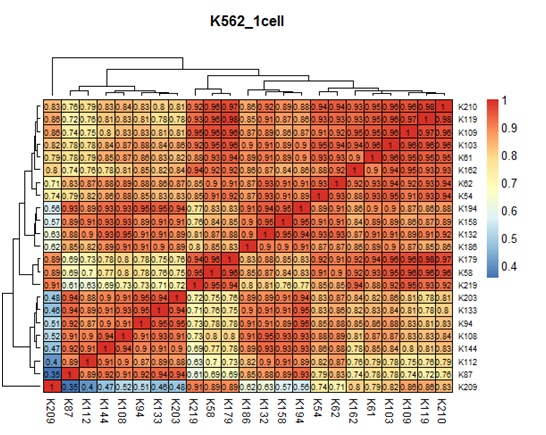
그림 5. Reproducibility of gene expression measurements.
SMARTer ICELL8 system으로 생성된 full-length RNA-seq 데이터의 재현성을 평가하기 위해, 확인 된 모든 transcript에 대한 유전자 발현 측정을 Heatmap으로 표시하였고, Heatmap에 표시된 것처럼 모든 single cell이 포함된 well 사이의 Pearson correlation를 결정하는데 사용하였다.