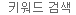- 고객지원
- 업무안내│제품문의
- 전화번호│02-2081-2510
- Email │support@takara.co.kr
- 업무시간안내
- [ 평 일 ] 09 : 00 ~ 18 : 00 │ [ 점심시간 ] 12 : 00 ~ 13 : 00
- 토·일요일, 공휴일은 휴무입니다.
Home >
전제품보기 >
기기 · 소모품 >
NGS 관련장비 >
High-throughput single-cell T-cell receptor profiling by SMART technology
High-throughput single-cell T-cell receptor profiling by SMART technology

- Confident clonotype calling
SMARTer ICELL8 workflow는 생성된 데이터에 대한 확신을 제공하는 positive control과 negane control을 사용이 가능합니다. - Sensitive detection
대다수의 read가 pairing information을 가지는 Full-length reads입니다. - Useful for complex samples
RACE 기술에 기초한 접근으로 낮은 empty도의 TCR variants의 검출도 가능합니다.
Introduction
벌크 샘플의 T-cell receptor (TCR) profiling은 TCR 레파토리 다positive, TCR 매개 항원 특이성 및 적응 면역 반응의 메커니즘에 대한 우리의 이해를 향상시켰다. 그러나 이러한 연구에서 발현되는 clonotype과 벌크 집단에서의 상대적 empty도를 알 수 있지만 아주 드문 cell 집단을 제외하고는 cell의 특정 receptor chain의 올바른 alpha - beta (αβ) pairing을 결정하는 것은 거의 불가능합니다.
Single-cell T-cell receptor (scTCR) clonotype 분석을 통해 각 cell에서 발현되는 특정 TCR αβ chain pairing의 결정이 가능합니다. 이러한 pairing 정보를 통해 연구자는 T-cell의 heterogenecity 및 plasticity에 대해 이해가 가능하고, 개별 TCR의 항원 특이성에 대한 pairing의 분포를 결정하고, 치료용 항체를 디자인할 수 있습니다. 여기서 우리는 TCR-α 및 TCR-β의 full-length에서의 variable region을 확보하기 위해 SMARTer ICELL8 Single-Cell System 또는 SMARTer ICELL8 cx Single-Cell System과 함께 5'-RACE 유사 접근법과 SMART 기술을 활용한 새로운 NGS (NGS) library preparation kit를 사용하였다. (TCR-α와 TCR-β는 발현된 단백질 사슬을 지칭하는데 사용되는 반면,이 기술 노트에서 TCR-a와 TCR-b는 nucleic acid transcript를 나타내기 위해 사용됩니다.) single cell 분리 및 nanowell chip에서 nano liter 스케일 PCR을 가능하게하는 human TCR a/b profiling workflow와 자동화 된 SMARTer ICELL8 플랫폼을 사용하면 1,000개 이상 cell의 clonotype 분석이 가능합니다. 이 workflow는 또한 증폭된 cDNA에서 5'-end capture를 통해 differential expression 데이터를 도출하는데 사용할 수도 있습니다.

그림 1. SMARTer ICELL8 workflow overview with safe process stopping points indicated.
1,728개의 고유한 바코드가 있는 template-switching oligos(TSO)가 SMARTer ICELL8 TCR chipchip에 3반복으로 분주되어 있고, cell은 well당 평균 1개의 cell로 TCR chip에 분주된다. CellSelect Software는 고유한 바코드가 있는 single cell이 들어있는 well을 식별합니다. cell lysis 후 이어지는 oligo-dT priming을 통해 cDNA 합성이 이뤄지고, 각 cell에서의 mRNA에 바코드합니다. Chip위에 well 특이적인 barcode와 template-switching oligos (TSOs)가 각각의 cell의 transcriptome을 정의하기 위해 RT-PCR에서 single primer로 사용됩니다.
증폭된 cDNA는 하나의 collection tube에 모아집니다. TCR variable region의 TCRa 및 TCRb 서열은 TCR subunit의 contant region을 타겟으로 하는 primer를 사용하여 pooling된 cDNA로부터 선택적으로 증폭된다. TCR 복합체중에서 TCRa 및 TCRb subunit에 대한 특정 amplicon은 gene specific primer(GSP)를 이용한 indexing된 nested PCR을 사용하여 만든다. 전체 workflow 시간은 hand-on time 2-3시간을 포함하여 2일정도 소요됩니다.
Results
TCR clonotype calls in different cell lines using the SMARTer ICELL8 workflow
아래에 설명된 실험에서 SMARTer ICELL8 Human TCR a/b profiling workflow는 SMARTer ICELL8 Single-Cell System에서 분리된 T-cell 및 말초 혈액 단핵 cell (PBMCs)를 처리하는데 사용되었습니다. cDNA는 chip상에서 수행된 RT-PCR동안 oligo-dT priming을 통해 생성되었다 (그림 2, 상단). chip에 인쇄된 well-specific 바코드가 있는 template-switching oligos는 single-primer amplification을 사용하여 모든 각각의 cell의 transcriptome을 정의하는데 사용되었습니다. chip으로부터 cDNA를 추출한 후, TCR variable region의 TCRa 및 TCRb 서열을 TCR subunit의 constnat region을 타겟으로하는 primer를 사용하여 pooling된 cDNA로부터 선택적으로 증폭시켰다. TCR 복합체의 TCRa 및 TCRb 서브 유닛을 위한 특정 amplicon은 gene specific primer(GSP; 그림 2, 하부)를 갖는 indexing된 nested PCR을 사용하여 만들었다.
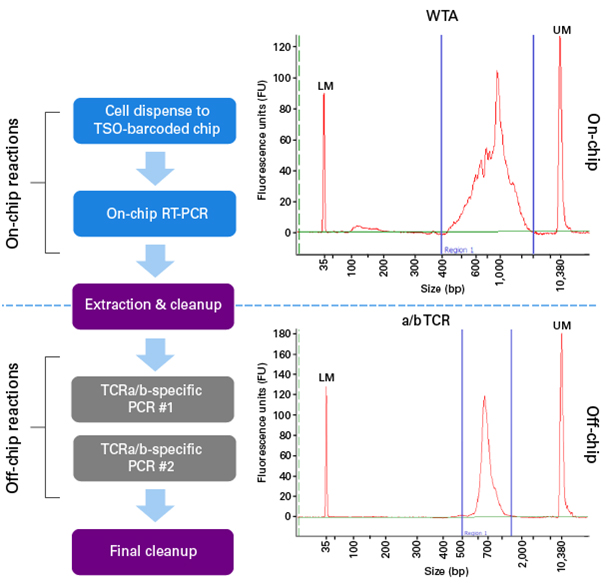
그림 2. TCR-seq workflow.
[상단] SMARTer ICELL8 TCR chip에 샘플(cell, positive control, negative control)를 분주한 후, oligo-dT priming chip을 통해 cDNA를 합성했습니다. 정제된 full-length cDNA에 대한 Bioanalyzer 결과,평균 cDNA amplicon 길이는 964bp임을 확인하였습니다. "LM"과 "UM"으로 표시된 peak는 DNA reference marker임.
[하부] Chip으로부터 cDNA를 추출한 후, TCR variable region의 TCRa 및 TCRb 서열을 선택적으로 증폭시켰다. SMARTer ICELL8 Human TCR a/b profiling workflow를 사용하여 생성된 TCRa 및 TCRb 서열을 포함하는 library의 Bioanalyzer trace는 650bp에서 1,150bp 사이의 넓은 피크와 700bp에서 900bp 범위에서 최대 피크를 보여줍니다. "LM"과 "UM"으로 표시된 peak는 DNA reference marker임.
SMARTer ICELL8 system의 이점은 데이터에 대한 신뢰를 높이기 위해 여러 컨트롤을 설정할 수 있다는 것입니다. 샘플을 제외한 모든 reaction component가 포함된 negative control을 사용하여 자신감있는 clonotype call에 대한 threshold를 설정할 수 있습니다. 총 15개의 negative 대조구가 이 실험에 포함되어 있으며, 확실한 clonotype call에 대한 threshold를 설정하기 위해 negative control well에서 상위 clonotype에 연관된 read수를 확인했습니다. 이러한 well은 비어 있기 때문에, negative control well과 연관된 모든 clonotype은 background이다. 우리는 이 empty well에 대한 평균 clonotype 판독 값에 3번 표준 편차를 더하여 clonotype 판독 수에 대한 threshold 값을 설정했습니다. 이 실험에서 threshold 값은 8개의 clonotype 판독 값으로 설정되었습니다 (그림 3, 녹색 컷 - 오프 라인). 이 threshold 값 미만의 판독 카운트로 모든 clonotype call을 제거한 후, 아미노산 서열을 조사하고 정지 코돈 또는 프레임 변이 돌연변이를 함유한 것을 제거하였다. Positive control은 Jurkat total RNA로 설정하였다. Jurkat RNA가 들어있는 well에 대한 clonotype call은 Jurkat TCRa 및 TCRb clonotypes에 특정한 read만을 보여주므로 well간의 barcode crosstalk이 없음을 나타냅니다.
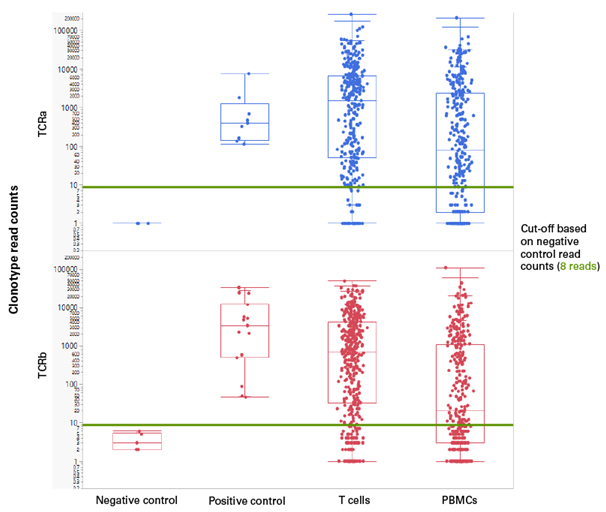
그림 3. Clonotype read counts for T cells and PBMCs.
MiXCR 결과를 Excel을 사용하여 필터링하여 1X PBS의 negative control보다 큰 TCRa 및 TCRb clonotype (실선 기호, 8 read count)에 대한 read threshold를 설정하였다. 데이터를 JMP 소프트웨어를 사용하여 분석하고 박스 플롯으로 나타내었다. 각 점은 cell에서 TCRa (청색) 및 TCRb (적색) clonotype을 나타냅니다. chip negative control은 샘플과 NTC 또는 낮은 read의 "junk" clonotype 사이의 구별을 가능하게 하는 threshold를 설정하는 데 사용되었습니다.
Assessment of alpha-beta chain pairing
다음으로 TCRa와 TCRb 서열이 검출된 cell를 결정하기 위해 데이터를 검사했다 (표 1). positive control 샘플의 경우, 모든 well은 TCRa 및/또는 TCRb에 대한 clonotype call을 포함하였다. PBMC 샘플의 경우 PBMC는 T-cell보다 많은 것으로 구성되었기 때문에 cell의 약 44 %가 TCRa 및/또는 TCRb에 대한 clonotype 호출이 있었다. T-cell 샘플의 약 75%는 TCRa 및 / 또는 TCRb에 대한 서열을 포함하였다. 이것은 T-cell 준비 중에 negative control 선택이 완료되지 않았기 때문에 일부 cell에는 검출 가능한 TCR transcripts가 없었을 가능성이 큽니다. 또한, 발현이 너무 낮아 일부 cell에 존재하는 TCR clonotype을 검출할 수 없었을 수 있다.
표 I. Percentage of samples with clonotype calls
데이터를 더 자세히 살펴보면, clonotype call을 포함하는 PBMCs와 T cell의 대다수는 alpha와 beta transcript를 가지고 있어 αβ pairing이 검출되었음을 나타냅니다 (그림 4). PBMCs의 경우, TCR transcript가 검출된 44%의 샘플 중 70%가 αβ pairing을 보였다. 한편, 검출된 TCR transcript를 갖는 T-cell의 63%는 αβ pairing을 함유 하였다. 흥미롭게도, pairing 결과의 대부분은 αß-only pairing 밖에 있었습니다. 예를 들어, beta-beta(ββ), alpha-alpha(αα), alpha-alpha-beta(ααβ) 및 기타 비표준 조합이 관찰되었습니다. 유사한 연구결과가 다른 연구들에서도 보고되었기 때문에 (Stubbington et al., 2016), 특이한 것으로 간주되지 않는다.
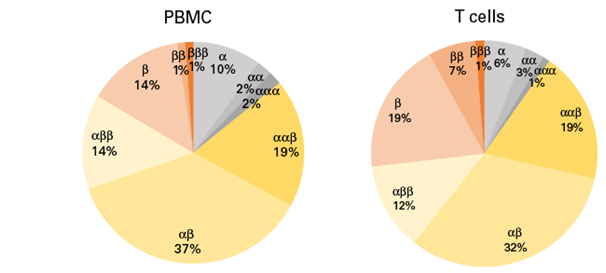
그림 4. Figure 4. Alpha and beta chain pairs are called in 70% of PBMCs and 63% of T cells with identified clonotypes.
scTCR Analyzer 파이프 라인의 출력 파일은 상위 세 가지 clonotype을 보고합니다. 분석 결과 αα, ββ, αββ, ααβ, ααα 및 βββ를 비롯한 여러 pairing 구성으로 나타났습니다.
TCRb transcript의 clonotype 분포
Sequencing 데이터에서 확인된 TCR clonotype의 분포는 코드 다이어그램을 사용하여 시각적으로 표시할 수도 있습니다 (그림 5). 코드 다이어그램는 분석된 PBMC 및 T cell에서 관찰된 TCRb clonotype 다양성을 나타냅니다. 두 샘플 모두에서 동일한 유형의 TRBJ 유전자 부위가 관찰되었다. 그러나 PBMCs에서 가장 높은 발현도를 보이는 clonotype은 TRBJ2-3 다음에 TRBJ2-7이었고 T cell에서는 TRBJ2-7이 가장 높게 발현되었고 TRBJ2-1이 뒤를 이었다. 유사하게, V 유전자 절편에 대해, 고도로 표현된 TRBV 유전자 절편은 두 가지 cell 유형에 대해 상이하다. PBMCs에서 TRBV5-1은 TRBV3-1이 뒤 따르는 가장 높은 발현을 보였다. T cell에서 TRBV20-1은 TRBV5-1에 이어 가장 높은 발현 clonotype이었다. 우리는 PBMC보다 T cell에서 더 많은 clonotype 다양성을 관찰했는데, 이는 399개 cell에서 유래한 T cell 데이터와 비교하여 261개 cell로부터 PBMC 데이터를 얻었기 때문으로 보인다. 전반적으로 이 데이터는 다양한 clonotype 조합을 확인하는 chemistry 반응의 민감도를 나타내므로 이 system은 복잡한 샘플을 보는 데 이상적인 선택이라고 보입니다.
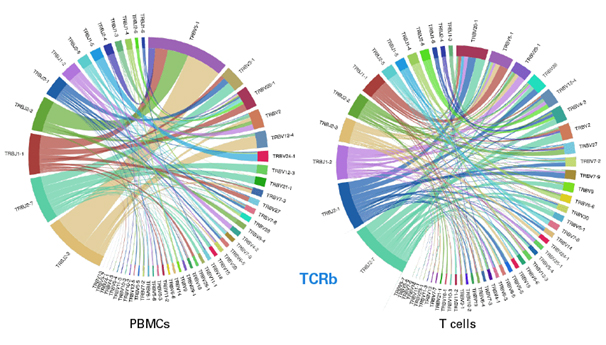
그림 5. Chord diagrams reveal TCRb clonotype distributions.
각 cell 유형 (왼쪽 : PBMCs, 오른쪽 : T cell)에 대한 코드 다이어그램은 표시된 TCRb Variable-Joining(V-J) segment 조합의 분포를 보여줍니다. 각 arc는 V 또는 J segment를 나타내며 segment가 데이터 세트에 표시되는 상대 비율에 따라 길이가 확장됩니다. 각 chord은 표시된 V-J 조합을 포함하고 데이터 집합에서 해당 조합의 상대적 존재량에 따라 가중치를 부여한 일련의 클론 형식을 나타냅니다.
Result
Human TCR a/b profiling workflow는 SMARTer ICELL8 system이 TCR αβ pairing 정보를결정하기 위해 수천 개의 single T cell로부터 Illumina sequencing library를 만드는 데 사용될 수 있습니다. TCR sequencing에 대한 매우 감도높은 접근법은 TCRa 및 TCRb variable region을 완전히 검출, 증폭하기 위해 SMART cDNA synthesis 및 RACE 기반의 gene-specific primingd을 이용한 TCR-specific PCR을 사용하여 이뤄집니다. 이러한 chemistry와 SMARTer ICELL8 single cell system 또는 SMARTer ICELL8 cx single의 결합은 쌍을 이루는 TCR clonotype의 다양성에 대한 연구에 대한이 접근법의 일반적인 유용성 및 확장성을 보여주는 1,000개 이상의 cell profiling을 가능하게 합니다. 또한 positive 및 negative control을 설정하는 능력은 특히 complex sample을 이용한 데이터에 대한 신뢰도를 높입니다.
Methods
TCRa 및 TCRb 시퀀스를 포함하는 library는 SMARTer ICELL8 Human TCR a/b 사용자 매뉴얼의 프로토콜에 따라 profiling workflow를 사용하여 생성되었습니다. positive control의 경우 Control Jurkat Total RNA (Takara Bio)를 사용하였다. 분리된 PBMCs (AllCells)를 RPMI에서 해동시키고 cell을 염색하기 전에 배지에서 한 번 씻어냈다. T cell를 EasySep Direct Human T cell 분리 키트 (STEMCELL Technologies)를 사용하여 권장 매뉴얼에 따라 전혈로부터 분리하였다. Sequencing을 위해 최종 library를 5% PhiX Control v3 (Illumina) spike in을 포함하여 13.5pM으로 희석했습니다. Sequencing은 600 Cycle MiSeq Reagent Kit v3 (Illumina)를 사용하여 2 x 300bp pairing 판독을 사용하여 Illumina MiSeq® sequencer에서 수행했습니다.
sequencing 후 각 pool에 대한 FASTQ 파일은 ICELL8 scTCR Analyzer를 사용하여 demultiplex하고 각 in-line index에 해당하는 샘플의 well에 read를 할당했습니다. 달리 언급되지 않는 한, 역 다중화는 인라인 인덱스의 정확한 일치를 사용하여 수행되었다. 각 샘플 well에 대한 레퍼토리 분석은 MiXCR 2.1.8 (Bolotin et al., 2015)을 사용하여 수행되었다. 읽기는 T cell receptor의 V, D, J 및 C 유전자를 기준으로 정렬한 다음 CDR3 유전자 region에 대한 clonotype 정보를 추출하여 일반 텍스트 파일에 보고했습니다. 그런 다음 개별 샘플 well 정보를 수집하는 전체 chip 요약 보고서가 생성되었습니다. 나중에 quality control tertiary 분석은 chip 내에 포함된 negative control (있는 경우)을 전제로 deletion 또는 frameshift 변이가 있는 clonotype을 제외하기 위해 실시되었다. Cell 유형별로 통계 및 clonotype 통계를 필터링하여 결과로 나타내었다.
References
Bolotin, D. A. et al. MiXCR: software for comprehensive adaptive immunity profiling. Nat. Methods 12, 380-1 (2015).
Stubbington, M. J. T. et al. T cell fate and clonality inference from single-cell transcriptomes. Nat. Methods 13, 329-32 (2016).
관련제품
제조사 |
제품코드 |
제품명 |
용량 |
가격 |
비고 |
사용자 |
TKR |
640179 |
SMARTer® ICELL8® Human TCR a/b Profiling - Indexing Primer Set |
1 Chip |
가격문의 |
||
TKR |
640180 |
SMARTer® ICELL8® Human TCR a/b Profiling - Indexing Primer Set |
5 Chips |
가격문의 |
|
|
TKR |
640181 |
SMARTer® ICELL8® Human TCR a/b Profiling - Indexing Primer Set |
10 Chip |
가격문의 |
|
|
TKR |
640182 |
SMARTer® ICELL8® Human TCR a/b Profiling Reagent Kit |
Each |
가격문의 |
|
|