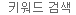- 고객지원
- 업무안내│제품문의
- 전화번호│02-2081-2510
- Email │support@takara.co.kr
- 업무시간안내
- [ 평 일 ] 09 : 00 ~ 18 : 00 │ [ 점심시간 ] 12 : 00 ~ 13 : 00
- 토·일요일, 공휴일은 휴무입니다.
Home >
전제품보기 >
기기 · 소모품 >
NGS 관련장비 >
A high-throughput method for single-cell ATAC-seq on the SMARTer ICELL8 Single-Cell System
A high-throughput method for single-cell ATAC-seq on the SMARTer ICELL8 Single-Cell System

Introduction
Epigenomic 정보는 유전자 조절에 대한 큰 통찰력을 제공하지만, 동일한 유형의 단일 세포라도 독특한 epigenomic profile을 가질 수 있기 때문에 매우 복잡합니다. 최근 몇 년 동안 single cell NGS 방법에 있어서 큰 진전이 이루어졌지만 염색질 상태를 검사하는 single cell 접근법은 매우 복잡하고 처리량이 낮으며 비용 또한 적지 않습니다. 최근 Stanford University의 Greenleaf 연구실은 Takara Bio USA, Inc.(TBUSA)와 공동으로 SMARTer ICELL8 Single-Cell System을 사용하여 single cell로부터 sequencing library를 preparation하는 매우 간단하고 신속한 ATAC-seq workflow를 개발했습니다.이 방법을 이용하면 chip상에서 workflow로 4~5시간 만에 1,000개 이상의 single cell에서 library를 제작할 수 있습니다.
Results
SMARTer ICELL8 system의 ATAC-seq workflow
SMARTer ICELL8 Single-Cell System은 single cell을 5,184-nanowell blank chip(그림 1)으로 자동 분리할 수 있습니다. Hoechst와 propidium iodide 염색법을 사용하여 cell viability을 분석하고 살아있는 single cell이 들어있는 well을 CellSelect Software를 사용하여 자동으로 식별하여 목적하는 well에만 시약을 분주합니다. Tagmentation, index 추가 및 PCR amplification이 chip상에서 (그림 1)에서 진행되므로 기술적 가변성을 최소화하고 4-5시간의 간단한 workflow를 제공합니다. 그런 다음 PCR amplicon을 chip에서 회수, pooling하여, size selection bead를 사용하여 sequencing library를 제작합니다. (자세한 프로토콜은 프로토콜 페이지를 참조하십시오).
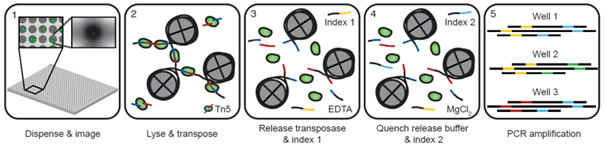
그림1. Schematic of the ATAC-seq workflow on the SMARTer ICELL8 system.
ATAC-seq reveals distinct cell populations based upon chromatin accessibility at transcription factor start sites
말초혈액 단핵세포(PBMC), B, CD4+T, CD8+T, monocyte 및 T 세포를 전혈로 부터 분리하였다. SMARTer ICELL8 시스템을 이용하여 Smartchip에 dispense하였습니다. QC를 통과한 총 2,333개의 single cell을 사용하여 cell당 평균 14,000개의 fragment로 매우 flexible한 single cell ATAC-seq library(그림 2)를 생성하고 sequencing하였다.
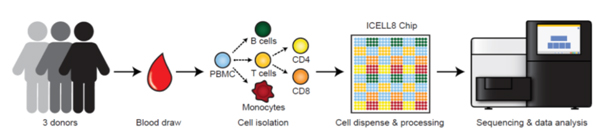
그림 2. Experimental design for using ATAC-seq to identify epigenomic states of multiple cell types from human donors.
전사 시작 부위 내 염색질 접근성 분석 결과 세 가지 주요 군집인 B세포, T세포 및 monocytes (그림 3)로의 매우 견고한 그룹화가 나타났습니다. PBMC subpopulation은 여러 분리된 세포 유형(그림 3, Panel A)과 일치하는 세포 유형별 염색질 접근성 패턴을 나타냈습니다. PU.1 DNA binding motif의 결과는 특히 monocytes 및 B cell에서의 특이성을 증가시키는 이전에 발표된 문헌과 일치하였으나(그림 3, Panel B), 반면에 C/EBPα 및 RUNX1 motif는 monocytes 및 T cell에서만 보다 접근하기 쉬웠다 (그림 3, Panel C, D).
그림 3. t-SNE plots of chromatin accessibility reveal robust clustering of chromatin accessibility patterns within specific hematopoietic cell types.
모든 세포 (Panel A), PU.1 (Panel B), C/EBPα (Panel C) 및 RUNX1 (Panel D)에 대한 chromatin 접근성 프로파일은 human blood에서 유도된 주요 세포 유형의 개별 클러스터링을 나타냅니다.
Conclusion
TBUSA와 함께 Greenleaf 연구실에서 개발한 이 프로토콜을 이용하면 single cell ATAC-seq용 library를 신속하게 제작할 수 있습니다. SMARTer ICELL8 single cell system은 이러한 workflow를 간소화할 수 있도록 automated cell isolation/imaging을 제공하며 4-5시간의 간편하고 신속한 on-chip workflow가 가능하게 합니다. 이러한 방법은 chip당 1,500개 이상의 single cells을, cell당 평균 14,000개 이상의 fragment를 single cell 분석이 가능하여, 이전에 설명한 염색질 접근성 프로파일을 반복하는 것과 아주 복잡한 샘플(예 : Human blood)에서도 특이적인 cell type을 확인하는 것이 가능합니다.
Method
상기 데이터의 도출에 사용된 full method는 Mezger et al., 2018에서 얻을 수 있지만 SMARTer ICELL8 single cell system에서 single cell ATAC-seq에 대한 사용가능한 프로토콜은 TBUSA에서 직접 얻을 수 있습니다.
References
Mezger, A. et al. High-throughput chromatin accessibility profiling at single-cell resolution. Nat. Commun. 9, 3647 (2018).