- 고객지원
- 업무안내│제품문의
- 전화번호│02-2081-2510
- Email │support@takara.co.kr
- 대전지사
- 업무안내│대전/충청지역 주문, 제품문의
- 전화번호│042-828-6525
- Email │tkbd@takara.co.kr
- 업무시간안내
- [ 평 일 ] 09 : 00 ~ 18 : 00 │ [ 점심시간 ] 12 : 00 ~ 13 : 00
- 토·일요일, 공휴일은 휴무입니다.
[Cancer Research] Cancer biomarker quantification


다양한 biomarker panel과 연구
High-throughput studies
몇몇 연구에서는 human oncology panel을 활용하여, 1,200개의 특이적 유전자를 분석하여 16 그룹으로 분류하였다. (signal transduction, cancer, apoptosis, angiogenesis, cardiovascular disease, ADME (toxicology, drug metabolism, and transport), inflammation, kinases, transcription factors, drug targets, G-protein coupled receptors, cell cycle and proliferation, DNA damage repair, growth factors, proteinases, phosphatases)
그 중 한 연구에서는 이 human oncology panel을 이용하여, human cytomegalovirus (HCMV)와 암과의 상호작용을 분석함으로써, 신호 전달 (MAPK, PI3K/Akt)과 감염, 혈관 신생, 종양 억제, DNA 손상 복구 경로 등을 포함한 다양한 경로에서 발현되는 20개의 유전자를 발견하였다. 이를 통해, HCMV가 건강한 정상 세포에서 암이나 종양 활성을 유발할 수 있다는 가능성을 확인하였다. 또 다른 연구에서는 동일한 panel을 이용해, 단백질 합성에 상호작용하는 인자인 tyrosyl-tRNA synthetase가 핵에 위치하여 DNA 손상 복구 유전자를 조절하는 방법에 대해 분석하였다.
다른 연구에서는 확인된 1,306개의 miRNA를 포함하는 miRNA panel과 SmartChip™ system을 이용하여, 환자로부터 얻은 수많은 대장암 세포와 췌장 관선암 세포 (pancreatic ductal adenocarcinoma cell) 샘플로부터 miRNA를 선별함으로써 질병에 관여하는 주요 miRNA 매개체를 확인하였다. 이 panel은 암 외에도 비침습적 샘플을 이용한 산전 유전자 검사에 활용될 수 있으며, 산모 혈장으로부터 다운 증후군과 연관된 miRNA를 확인하는데 적용된 바 있다. 또한, miRNA panel은 각기 다른 세포 집단를 연구하고 특징을 확인하는데 이용할 수 있어, 한 연구에서는 암세포와 줄기세포 간의 세포 사멸을 조절하는 공통된 miRNA를 확인하였다. 이뿐만 아니라, 한 연구에서는 혈청, 혈장, 백혈구에서의 miRNA 하위 그룹에 대한 특징을 분석하였으며, 다른 연구에서는 SmartChip™ system을 이용하여 miRNA-98에 의존하는 T 세포의 glucocorticoid 억제 작용에 대해 확인하였다.
SmartChip™ system에서 panel을 이용해 1,700개의 assay로 IncRNA를 연구한 경우도 있다. 이 panel은 Biogazelle와 공동으로 개발되었으며, 게놈 데이터베이스와 대조해, Minimum Information for Publication of Quantitative Real-Time PCR Experiments (MIQE) 가이드라인에 준수하여 제작되었다. 한 연구에서는 이 IncRNA panel을 이용하여, 뇌, 혈액, 골수, 유방, 장, 신장, 자궁, 전립선, 피부를 포함한 다양한 조직으로부터 파생된 60개의 인간 암 세포주인 NCI-60 암세포주의 발현을 분석하였다. 1,700개의 IncRNA 중 97%가 NCI-60 암세포주에서 재현성 있게 발현됨을 이 panel으로 확인하였다. 또 다른 연구에서는 IncRNA panel을 이용하여, 흑색종에서의 흑색종을 생성하는 주요 매개체에 대해 분석함으로써, IncRNA SAMMSON이 흑색종을 고도로 발현시키는 것을 확인하였고 그에 대한 작용과 후속 실험을 진행했다. 최근에 진행된 한 연구에서는 somatic copy-number alterations (SCNA)가 암유전자 혹은 종양 억제 유전자와 같은 역할을 하는 IncRNA alternation에 미치는 영향을 확인하기 위해 IncRNA panel을 개발하였으며, 암을 유발하는 IncRNA 확인하는 데 이를 활용할 수 있다.
Better biobanking with high-throughput qPCR
FFPE 방법을 이용하면, 생물학적 샘플을 상온에서 오랜 시간 쉽게 보관할 수 있으나, 오랜 고정 시간으로 인해, assay를 통한 검출이 어려울 정도로 핵산이 손상될 수 있다는 단점이 있다. 이 한계를 극복하기 위해 다카라바이오가 지원하는 대량 qPCR 분석이 가능한 SmartChip™ system을 적용할 수 있다. 본 제품을 이용하면 상태가 좋은 샘플뿐 만 아니라 FFPE, 동결 보존된 조직을 대량으로 qPCR 분석하고자 할 때 각각의 platform을 최적화하고, hands-on time과 시약 사용량을 모두 최소화하여 분석할 수 있다 (그림1).
Integrated Biobank of Luxembourg (IBBL)의 연구자들은 이를 이용한 FFPE 샘플의 분석법에 대한 연구를 진행한 바 있다 (Ammerlaan, W. et al.). Biobank에서는 다양한 장기와 질병에 대한 수많은 FFPE 샘플에 대한 연구가 진행될 수 있도록 지원하고 있으나, 샘플의 고정 시간이 오래됨에 따라 핵산이 손상되어 연구에 어려움이 있었다. 이를 위해 IBBL 연구자들은 6, 12, 23, 72시간동안 고정된 신장과 자궁 FFPE 샘플을 이용해 miRNA와 small nucleolar RNA (snoRNA)의 발현을 확인하고자 했다.
Small RNA의 타겟이 굉장히 많기 때문에, IBBL 연구자들은 1,000개 이상의 miRNA와 snoRNA 타겟을 분석할 수 있는 SmartChip™ Human miRNA Panel v3.0을 SmartChip™ system에 적용하여 분석하였다. 논문의 저자는 각각의 chip 3개로 reference human total RNA를 분석하였으며, 각각을 비교하여 R2값이 0.98 이상을 얻어 실험 재현성을 확인하였다. 이 후, 신장과 자궁 샘플을 SmartChip™ Human miRNA Panel v3.0에서 분석하여 고정 조건에 따른 miRNA, snoRNA 발현 차이를 비교하였으며, snoRNA에서 발현이 안정적이지 않음을 확인하였다. 연구자들은 SCARNA5, SNORA16A, SNORA44, SNORA61, 총 4개의 snoRNA를 이용하여, “snoRNA CNRQ score”를 개발하였으며, 이를 활용하면 6-12시간 혹은 72시간 고정된 샘플을 구분할 수 있다.
다만, 이 방법은 SmartChip™ system에서 단일 샘플을 분석하는 경우에만 적용할 수 있기에, 한계를 극복하기 위해 “snoRNA score”을 개발하였다. 이를 이용하면, 1,000번 이상의 분석이 필요한 SmartChip™ Human miRNA Panel v3.0 대신, 고정 시간에 따라 값이 변화하는 snoRNA와 고정 시간과 관계없이 값이 일정한 miRNA control을 포함하는 24개의 타겟만으로도 분석이 가능하다. IBBL 연구자들은 보다 많은 처리량을 분석하기 위해 “snoRNA score”을 SmartChip™ system에 적용시켰고, 40개의 샘플을 정확하게 분석할 수 있었다.
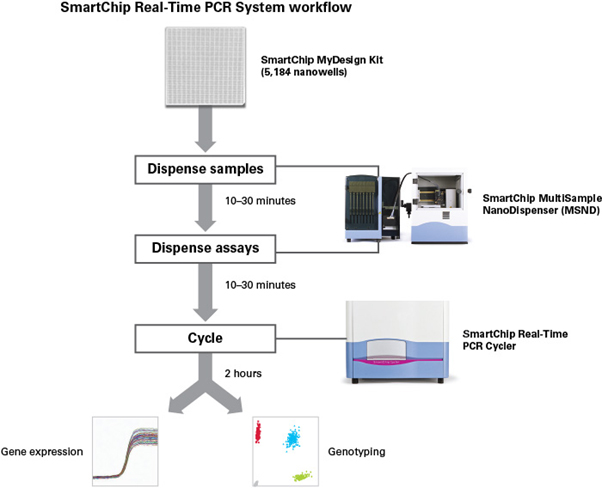
그림 1. Overview of the SmartChip™ real-time PCR system.
[원문] Using mRNA, miRNA, and lncRNA as biomarkers for cancer and other diseases
[참고문헌]
- Ammerlaan, W. et al. Small Nucleolar RNA Score: An Assay to Detect Formalin-Overfixed Tissue. Biopreserv. Biobank. 16, 467 (2018).
- Ammerlaan, W. & Betsou, F. Intraindividual Temporal miRNA Variability in Serum, Plasma, and White Blood Cell Subpopulations. Biopreserv. Biobank. 14:bio.2015.0125 (2016).
- Chen, X. et al. Defining a population of stem-like human prostate cancer cells that can generate and propagate castration-resistant prostate cancer. Clin. Cancer Res. 22:4505-4516 (2016).
- Choo, K. B., Soon, Y. L., Nguyen, P. N. N., Hiew, M. S. Y. & Huang, C.-J. MicroRNA-5p and -3p co-expression and cross-targeting in colon cancer cells. J. Biomed. Sci. 21:95 (2014).
- Davis, T. E., Kis-Toth, K., Szanto, A. & Tsokos, G. C. Glucocorticoids suppress T cell function by up-regulating microRNA-98. Arthritis Rheum. 65:1882-1890 (2013).
- Ding, N. et al. BRD4 is a novel therapeutic target for liver fibrosis. Proc. Natl. Acad. Sci. U. S. A. 112:15713-8 (2015).
- Kamhieh-Milz, J. et al. Differentially expressed microRNAs in maternal plasma for the noninvasive prenatal diagnosis of down syndrome (Trisomy 21). Biomed Res. Int. 2014 (2014).
- Leucci, E. et al. Melanoma addiction to the long non-coding RNA SAMMSON. Nature 531:518-522 (2016).
- Mestdagh, P. et al. Evaluation of quantitative miRNA expression platforms in the microRNA quality control (miRQC) study. Nat. Methods 11:809-815 (2014).
- Mestdagh, P., Lefever, S., Volders, P. & Derveaux, S. Long non-coding RNA expression pro fi ling in the NCI 60 cancer cell line panel using. Sci. Data 1-6 doi:10.1038/sdata.2016.52 (2016).
- Nault, R., Fader, K. A. & Zacharewski, T. RNA-Seq versus oligonucleotide array assessment of dose-dependent TCDD-elicited hepatic gene expression in mice. BMC Genomics 16:373 (2015).
- Nguyen, P. N. N., Huang, C.-J., Sugii, S., Cheong, S. K. & Choo, K. B. Selective activation of miRNAs of the primate-specific chromosome 19 miRNA cluster (C19MC) in cancer and stem cells and possible contribution to regulation of apoptosis. J. Biomed. Sci. 24:20 (2017).
- Rothwell, D. G. et al. Evaluation and validation of a robust single cell RNA-amplification protocol through transcriptional profiling of enriched lung cancer initiating cells. BMC Genomics 15:1129 (2014).
- Sardi, S. H., Glassner, B. J., Chang, J. R. & Yim, S. H. Identification of Critical Biomarkers Responsive to Anti-Autophagy Therapies for Pancreatic Ductal Adenocarcinoma through a Performance Analysis of miRNA Platforms. J. Bioanal. Biomed. 1:doi:10.4172/1948-593X.S10-001 (2014).
- Snyder, C. et al. Prevalence of PALB2 mutations in the Creighton University Breast Cancer Family Registry. Breast Cancer Res. Treat. 150:637-641 (2015).
- Ullmann, P. et al. Hypoxia-responsive miR-210 promotes self-renewal capacity of colon tumor-initiating cells by repressing ISCU and by inducing lactate production. Oncotarget. 7 (2016).
- Volders, P.-J. et al. Targeted genomic screen reveals focal long non-coding RNA copy number alterations in cancer. bioRxiv 113316:doi:10.1101/113316 (2017).
- Wei, N. et al. Oxidative stress diverts trna synthetase to nucleus for protection against dna damage. Mol. Cell 56:323-332 (2014).
- Wu, L. et al. Full-length single-cell RNA-seq applied to a viral human cancer: applications to HPV expression and splicing analysis in HeLa S3 cells. Gigascience 4:51 (2015).

많은 타겟을 빠르고 민감하게 분석하기 위해, 다카라바이오는 대량 (High-throughput) qPCR을 위한
SmartChip™ System을 지원하고 있다.
본 제품은 다양한 연구에서 각각의 질병이 가지는 고유한 biomarker와 그 변화를 대량 분석하는데 이용되었으며, 이는 mRNA, miRNA, IncRNA 분석용 panel을 디자인하고 활용할 수 있다.
본 제품은 다양한 연구에서 각각의 질병이 가지는 고유한 biomarker와 그 변화를 대량 분석하는데 이용되었으며, 이는 mRNA, miRNA, IncRNA 분석용 panel을 디자인하고 활용할 수 있다.
High-throughput studies
몇몇 연구에서는 human oncology panel을 활용하여, 1,200개의 특이적 유전자를 분석하여 16 그룹으로 분류하였다. (signal transduction, cancer, apoptosis, angiogenesis, cardiovascular disease, ADME (toxicology, drug metabolism, and transport), inflammation, kinases, transcription factors, drug targets, G-protein coupled receptors, cell cycle and proliferation, DNA damage repair, growth factors, proteinases, phosphatases)
그 중 한 연구에서는 이 human oncology panel을 이용하여, human cytomegalovirus (HCMV)와 암과의 상호작용을 분석함으로써, 신호 전달 (MAPK, PI3K/Akt)과 감염, 혈관 신생, 종양 억제, DNA 손상 복구 경로 등을 포함한 다양한 경로에서 발현되는 20개의 유전자를 발견하였다. 이를 통해, HCMV가 건강한 정상 세포에서 암이나 종양 활성을 유발할 수 있다는 가능성을 확인하였다. 또 다른 연구에서는 동일한 panel을 이용해, 단백질 합성에 상호작용하는 인자인 tyrosyl-tRNA synthetase가 핵에 위치하여 DNA 손상 복구 유전자를 조절하는 방법에 대해 분석하였다.
다른 연구에서는 확인된 1,306개의 miRNA를 포함하는 miRNA panel과 SmartChip™ system을 이용하여, 환자로부터 얻은 수많은 대장암 세포와 췌장 관선암 세포 (pancreatic ductal adenocarcinoma cell) 샘플로부터 miRNA를 선별함으로써 질병에 관여하는 주요 miRNA 매개체를 확인하였다. 이 panel은 암 외에도 비침습적 샘플을 이용한 산전 유전자 검사에 활용될 수 있으며, 산모 혈장으로부터 다운 증후군과 연관된 miRNA를 확인하는데 적용된 바 있다. 또한, miRNA panel은 각기 다른 세포 집단를 연구하고 특징을 확인하는데 이용할 수 있어, 한 연구에서는 암세포와 줄기세포 간의 세포 사멸을 조절하는 공통된 miRNA를 확인하였다. 이뿐만 아니라, 한 연구에서는 혈청, 혈장, 백혈구에서의 miRNA 하위 그룹에 대한 특징을 분석하였으며, 다른 연구에서는 SmartChip™ system을 이용하여 miRNA-98에 의존하는 T 세포의 glucocorticoid 억제 작용에 대해 확인하였다.
SmartChip™ system에서 panel을 이용해 1,700개의 assay로 IncRNA를 연구한 경우도 있다. 이 panel은 Biogazelle와 공동으로 개발되었으며, 게놈 데이터베이스와 대조해, Minimum Information for Publication of Quantitative Real-Time PCR Experiments (MIQE) 가이드라인에 준수하여 제작되었다. 한 연구에서는 이 IncRNA panel을 이용하여, 뇌, 혈액, 골수, 유방, 장, 신장, 자궁, 전립선, 피부를 포함한 다양한 조직으로부터 파생된 60개의 인간 암 세포주인 NCI-60 암세포주의 발현을 분석하였다. 1,700개의 IncRNA 중 97%가 NCI-60 암세포주에서 재현성 있게 발현됨을 이 panel으로 확인하였다. 또 다른 연구에서는 IncRNA panel을 이용하여, 흑색종에서의 흑색종을 생성하는 주요 매개체에 대해 분석함으로써, IncRNA SAMMSON이 흑색종을 고도로 발현시키는 것을 확인하였고 그에 대한 작용과 후속 실험을 진행했다. 최근에 진행된 한 연구에서는 somatic copy-number alterations (SCNA)가 암유전자 혹은 종양 억제 유전자와 같은 역할을 하는 IncRNA alternation에 미치는 영향을 확인하기 위해 IncRNA panel을 개발하였으며, 암을 유발하는 IncRNA 확인하는 데 이를 활용할 수 있다.
Better biobanking with high-throughput qPCR
FFPE 방법을 이용하면, 생물학적 샘플을 상온에서 오랜 시간 쉽게 보관할 수 있으나, 오랜 고정 시간으로 인해, assay를 통한 검출이 어려울 정도로 핵산이 손상될 수 있다는 단점이 있다. 이 한계를 극복하기 위해 다카라바이오가 지원하는 대량 qPCR 분석이 가능한 SmartChip™ system을 적용할 수 있다. 본 제품을 이용하면 상태가 좋은 샘플뿐 만 아니라 FFPE, 동결 보존된 조직을 대량으로 qPCR 분석하고자 할 때 각각의 platform을 최적화하고, hands-on time과 시약 사용량을 모두 최소화하여 분석할 수 있다 (그림1).
Integrated Biobank of Luxembourg (IBBL)의 연구자들은 이를 이용한 FFPE 샘플의 분석법에 대한 연구를 진행한 바 있다 (Ammerlaan, W. et al.). Biobank에서는 다양한 장기와 질병에 대한 수많은 FFPE 샘플에 대한 연구가 진행될 수 있도록 지원하고 있으나, 샘플의 고정 시간이 오래됨에 따라 핵산이 손상되어 연구에 어려움이 있었다. 이를 위해 IBBL 연구자들은 6, 12, 23, 72시간동안 고정된 신장과 자궁 FFPE 샘플을 이용해 miRNA와 small nucleolar RNA (snoRNA)의 발현을 확인하고자 했다.
Small RNA의 타겟이 굉장히 많기 때문에, IBBL 연구자들은 1,000개 이상의 miRNA와 snoRNA 타겟을 분석할 수 있는 SmartChip™ Human miRNA Panel v3.0을 SmartChip™ system에 적용하여 분석하였다. 논문의 저자는 각각의 chip 3개로 reference human total RNA를 분석하였으며, 각각을 비교하여 R2값이 0.98 이상을 얻어 실험 재현성을 확인하였다. 이 후, 신장과 자궁 샘플을 SmartChip™ Human miRNA Panel v3.0에서 분석하여 고정 조건에 따른 miRNA, snoRNA 발현 차이를 비교하였으며, snoRNA에서 발현이 안정적이지 않음을 확인하였다. 연구자들은 SCARNA5, SNORA16A, SNORA44, SNORA61, 총 4개의 snoRNA를 이용하여, “snoRNA CNRQ score”를 개발하였으며, 이를 활용하면 6-12시간 혹은 72시간 고정된 샘플을 구분할 수 있다.
다만, 이 방법은 SmartChip™ system에서 단일 샘플을 분석하는 경우에만 적용할 수 있기에, 한계를 극복하기 위해 “snoRNA score”을 개발하였다. 이를 이용하면, 1,000번 이상의 분석이 필요한 SmartChip™ Human miRNA Panel v3.0 대신, 고정 시간에 따라 값이 변화하는 snoRNA와 고정 시간과 관계없이 값이 일정한 miRNA control을 포함하는 24개의 타겟만으로도 분석이 가능하다. IBBL 연구자들은 보다 많은 처리량을 분석하기 위해 “snoRNA score”을 SmartChip™ system에 적용시켰고, 40개의 샘플을 정확하게 분석할 수 있었다.

그림 1. Overview of the SmartChip™ real-time PCR system.
Code |
제품명 |
용량 |
640022 |
1 대 |
[원문] Using mRNA, miRNA, and lncRNA as biomarkers for cancer and other diseases
[참고문헌]
- Ammerlaan, W. et al. Small Nucleolar RNA Score: An Assay to Detect Formalin-Overfixed Tissue. Biopreserv. Biobank. 16, 467 (2018).
- Ammerlaan, W. & Betsou, F. Intraindividual Temporal miRNA Variability in Serum, Plasma, and White Blood Cell Subpopulations. Biopreserv. Biobank. 14:bio.2015.0125 (2016).
- Chen, X. et al. Defining a population of stem-like human prostate cancer cells that can generate and propagate castration-resistant prostate cancer. Clin. Cancer Res. 22:4505-4516 (2016).
- Choo, K. B., Soon, Y. L., Nguyen, P. N. N., Hiew, M. S. Y. & Huang, C.-J. MicroRNA-5p and -3p co-expression and cross-targeting in colon cancer cells. J. Biomed. Sci. 21:95 (2014).
- Davis, T. E., Kis-Toth, K., Szanto, A. & Tsokos, G. C. Glucocorticoids suppress T cell function by up-regulating microRNA-98. Arthritis Rheum. 65:1882-1890 (2013).
- Ding, N. et al. BRD4 is a novel therapeutic target for liver fibrosis. Proc. Natl. Acad. Sci. U. S. A. 112:15713-8 (2015).
- Kamhieh-Milz, J. et al. Differentially expressed microRNAs in maternal plasma for the noninvasive prenatal diagnosis of down syndrome (Trisomy 21). Biomed Res. Int. 2014 (2014).
- Leucci, E. et al. Melanoma addiction to the long non-coding RNA SAMMSON. Nature 531:518-522 (2016).
- Mestdagh, P. et al. Evaluation of quantitative miRNA expression platforms in the microRNA quality control (miRQC) study. Nat. Methods 11:809-815 (2014).
- Mestdagh, P., Lefever, S., Volders, P. & Derveaux, S. Long non-coding RNA expression pro fi ling in the NCI 60 cancer cell line panel using. Sci. Data 1-6 doi:10.1038/sdata.2016.52 (2016).
- Nault, R., Fader, K. A. & Zacharewski, T. RNA-Seq versus oligonucleotide array assessment of dose-dependent TCDD-elicited hepatic gene expression in mice. BMC Genomics 16:373 (2015).
- Nguyen, P. N. N., Huang, C.-J., Sugii, S., Cheong, S. K. & Choo, K. B. Selective activation of miRNAs of the primate-specific chromosome 19 miRNA cluster (C19MC) in cancer and stem cells and possible contribution to regulation of apoptosis. J. Biomed. Sci. 24:20 (2017).
- Rothwell, D. G. et al. Evaluation and validation of a robust single cell RNA-amplification protocol through transcriptional profiling of enriched lung cancer initiating cells. BMC Genomics 15:1129 (2014).
- Sardi, S. H., Glassner, B. J., Chang, J. R. & Yim, S. H. Identification of Critical Biomarkers Responsive to Anti-Autophagy Therapies for Pancreatic Ductal Adenocarcinoma through a Performance Analysis of miRNA Platforms. J. Bioanal. Biomed. 1:doi:10.4172/1948-593X.S10-001 (2014).
- Snyder, C. et al. Prevalence of PALB2 mutations in the Creighton University Breast Cancer Family Registry. Breast Cancer Res. Treat. 150:637-641 (2015).
- Ullmann, P. et al. Hypoxia-responsive miR-210 promotes self-renewal capacity of colon tumor-initiating cells by repressing ISCU and by inducing lactate production. Oncotarget. 7 (2016).
- Volders, P.-J. et al. Targeted genomic screen reveals focal long non-coding RNA copy number alterations in cancer. bioRxiv 113316:doi:10.1101/113316 (2017).
- Wei, N. et al. Oxidative stress diverts trna synthetase to nucleus for protection against dna damage. Mol. Cell 56:323-332 (2014).
- Wu, L. et al. Full-length single-cell RNA-seq applied to a viral human cancer: applications to HPV expression and splicing analysis in HeLa S3 cells. Gigascience 4:51 (2015).