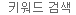- 고객지원
- 업무안내│제품문의
- 전화번호│02-2081-2510
- Email │support@takara.co.kr
- 대전지사
- 업무안내│대전/충청지역 주문, 제품문의
- 전화번호│042-828-6525
- Email │tkbd@takara.co.kr
- 업무시간안내
- [ 평 일 ] 09 : 00 ~ 18 : 00 │ [ 점심시간 ] 12 : 00 ~ 13 : 00
- 토·일요일, 공휴일은 휴무입니다.
[Cancer Research] Gene editing


Performing a phenotypic screen using the Guide-it™ CRISPR Genome-Wide sgRNA Library System
최근, CRISPR/Cas9을 이용해 Whole genome sequencing (WGS)이 가능하다는 것이 확인되면서, 연구자들은 관심이 있는 생물학적 경로에 관련되어 있는 새로운 유전자를 빠르게 식별할 수 있게 되었다. RNAi를 이용한 knockdown과 같은 기존의 방법에 비해 CRISPR/Cas9을 이용한 스크리닝은 보다 확실한 knockout을 이용하여 목적 세포의 표현형을 더 쉽게 검출하고 분석할 수 있다. 이 효과를 극대화하기 위해서는, 사용하기 편리한 시스템을 이용해 인간 세포에 genome-wide phenotypic 스크리닝를 수행할 수 있어야 한다. 다카라바이오에서는 최근의 연구에 기반한 sgRNA library와 Lentivirus system을 접목하여 Guide-it™ CRISPR Genome-Wide sgRNA Library System를 개발하였다.
Library design features
Guide it™ CRISPR/Cas9을 이용한 표현형 스크리닝은 새로운 유전자를 식별하는데 있어, 그 효율을 좌우하는 모든 중요한 프로세스를 제공하고 있다. 제품 내에는 최적화된 sgRNA가 library 형태로 제공될 뿐 아니라 plasmid library 증폭의 최적화, Lentiviral packaging, 목적 세포로의 감염 과정까지 포함하여 구성되어 있다.
본 Library는 19,000개 이상의 단백질 코딩 유전자를 타겟으로, 각 유전자를 타겟으로 하는 sgRNA가 4개씩, 즉 총 76,000개 이상의 sgRNA로 구성되어 있다 (Doench et al. 2016). 더 많은 종류의 sgRNA를 포함한 library를 적용할 수는 있으나, sgRNA의 수가 많아지면 증폭 과정에서 재현성이 감소할 수 있다. 이러한 이유로 multiple plasmid amplification, Lentiviral packaging, target-cell transduction step을 순차적으로 진행하는 다른 library에서는 편향성이 높거나 sgRNA 재현성이 낮은 경우를 확인할 수 있다.
당사의 Library는 Brunello library (Doench et al. 2016)에 기반하여, 다른 library보다 유사한 유전자를 타겟으로 작용하는 등의 off-target effect가 낮고, 목적 유전자에 대한 특이성이 높은 sgRNA를 포함하고 있다.
스크리닝 과정은 어떤 성능의 Library를 선택하는 지가 sgRNA의 재현성을 좌우한다. 이상적인 Library는 각각의 sgRNA가 모두 정확히 동일한 양을 포함하고 있어야 하지만, 이는 sgRNA 합성과 plasmid library 증폭 과정으로 인해 현실적으로 불가능하다. 다카라바이오는 수십년 간 노하우를 바탕으로 cDNA library를 합성하고 증폭하는 과정을 최적화하여, Guide-it™ CRISPR Genome-Wide sgRNA Library System 내 sgRNA library를 굉장히 유사한 정도로 합성하고 증폭할 수 있도록 하였다. 각 lot의 library 내 모든 sgRNA의 90% 이상이 10배 이내의 분포 범위에 위치함을 확인하였다 (그림 1, Panel A).
본 제품은 Lentivirus 생산과 목적 세포로의 감염을 간소화할 수 있는 손쉬운 형태로 library를 제공함으로써, 사용자가 최적화를 진행하지 않고도 형질 도입된 목적 세포에서 sgRNA가 잘 작용할 수 있게 하였다 (그림 1, Panel B).
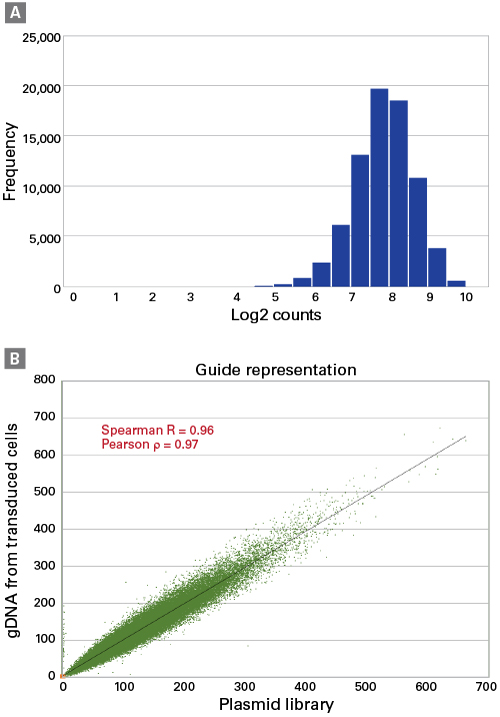
그림 1. Guide-it™ CRISPR Genome-Wide sgRNA Library System의 sgRNA 재현성 확인
(Panel A) sgRNA representation in the starting plasmid population. The Brunello-based sgRNA library was cloned into the pLVXS-sgRNA-mCherry-hyg Vector and amplified. Representation of the sgRNAs within the plasmid DNA was verified by next-generation sequencing (NGS). Bars represent the number of sgRNAs detected at a given read count within the population. (더 많은 sgRNA representation data를 확인하시려면 클릭하세요)
(Panel B) The correlation between read counts of each integrated sgRNA for the transduced cell population relative to read counts of the corresponding sgRNA for the starting plasmid population. These data show strong Spearman and Pearson correlations, indicating that the Guide-it CRISPR Genome-Wide sgRNA Library System allows for sgRNA representation to be maintained throughout transduction and selection of the target cell population.
Optimized lentiviral sgRNA library vector and sgRNA scaffold design for high editing efficiency
본 제품의 sgRNA library vector로는 pLVXS-sgRNA-mCherry-hyg이 사용 되었고, 성능과 안전성을 모두 고려하여 최적화했다 (그림 2, Panel A). 사용된 Lentiviral vector는 Cas9과 sgRNA를 감염 세포에서 각각 puromycin과 hygromycin로 선별 배양하게 된다. sgRNA library vector는 mCherry 단백질을 형광 리포터로 발현하고 있어, 형광 현미경에서 바로 도입 여부를 판별하고 역가를 확인할 수 있을 뿐 아니라, Cas9을 발현하는 세포주에 sgRNA library를 도입하기 위한 최적의 MOI (multiplicity-of-infection)를 보다 쉽게 결정할 수 있다 (그림 3). MOI는 각 Cas9 발현 세포에서 오직 하나의 sgRNA를 발현할 수 있도록 조절해야 하기에 아주 중요하다.
pLVXS-sgRNA-mCherry-hyg내의 sgRNA scaffold는 유전자 편집 효율을 향상시키기 위해 최적화된 서열로 구성되었으며 (그림 2, Panel B), sgRNA와 Cas9 endonuclease 간의 친화력을 극대화 할 수 있도록 디자인되었다 (Chen et al. 2013). Cas9과 sgRNA vector는 모두 다카라바이오의 Lenti-X™ Packaging Single Shots과 Xfect™ Transfection Reagent와 premix되어 동결 건조 형태로 제공되며, 물을 추가하여 Lenti-X™ 293T Producer Cell Line에 첨가하는 것 만으로 transfection을 위한 준비 과정이 완료된다.
그림 2. Guide-it™ CRISPR sgRNA library에 사용된 vector와 sgRNA scaffold
(Panel A) pLVXS-EF1a-Cas9-PGK-Puro and pLVXS-sgRNA-mCherry-hyg vector maps showing the lentiviral vector backbone. The vectors are self-inactivating for increased safety during production and use. The pLVXS-sgRNA-mCherry-hyg vector contains both mCherry and hygromycin markers expressed from an IRES-linked bicistronic expression cassette.
(Panel B) The sgRNAs, derived from the Brunello library, are expressed from the human U6 promoter and use an optimized scaffold sequence for better Cas9 loading and editing efficiency.
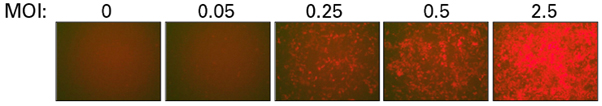
그림 3. mCherry 단백질을 형광 리포터로 이용한 sgRNA library Lentivirus의 도입 효율 확인
sgRNA Library-containing lentivirus was produced following the instructions in the user manual: water was added to a tube of sgRNA Library Transfection Mix, vortexed, incubated for 10 min and then added to the provided Lenti-X 293T cells. Lentivirus was harvested after 48 hours and used to transduce Cas9-expressing A375 cells at varying MOIs. Transduced cells were plated and analyzed for transduction efficiency by fluorescence microscopy after 48 hours.
Results - Editing activity of the Guide-it™ CRISPR Genome-Wide sgRNA Library in randomly selected clones
당사의 sgRNA library system의 편집 효율을 확인하기 위해, 흑색종 세포인 A375 세포에 Cas9과 sgRNA library를 형질 도입하여, 무작위로 20종의 clone을 선택하여 분석하였다. 각 Clone으로부터 sgRNA를 증폭하여 서열을 분석한 후, sgRNA가 타겟으로 하는 유전자에 대한 sgRNA primer를 디자인하였다 (그림 4, Panel A). 편집 효율은 Resolvase에 기반한 Guide-it™ Mutation Detection Kit을 이용하여 확인하였다 (그림 4, Panel B). 모든 개체에서 절단된 것이 확인되었으며, 이를 통해 성공적으로 유전자가 편집 되었음을 확인하였다. 각각의 clone에서 확인된 sgRNA는 실제의 plasmid sgRNA pool과 형질 도입된 세포의 gDNA로 분석한 correlation plot에 mapping 함으로써, 상대적인 read count를 확인하였으며, 각각의 sgRNA는 무작위로 선별되었음에도 plot 분포 내에 위치함이 확인되었다 (그림 4, Panel C).
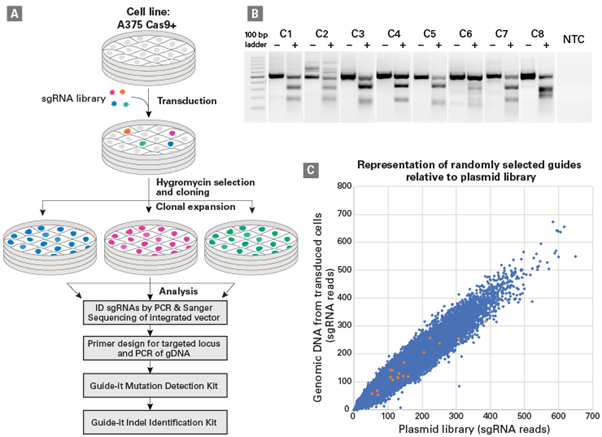
그림 4. 무작위로 선별된 개체에서 Guide-it™ CRISPR Genome-Wide sgRNA Library System의 편집 효율 확인
(Panel A) After transduction of Cas9+ A375 cells with the sgRNA library and hygromycin selection, twenty clones were selected randomly and expanded for analysis.
(Panel B) Genomic DNA was isolated from each clone and analyzed for activity using the Guide-it Mutation Detection Kit. The results of the resolvase assay for 8 representative clones are shown; NTC: non-transduced cells, (+) PCR products treated with Guide-it Resolvase, (-) untreated PCR products.
(Panel C) The sgRNA sequences from the twenty clones were mapped against the correlation plot of the plasmid library and transduced gDNA. The sgRNAs isolated from the randomly-selected clones are marked with orange dots within the correlation plot of all sgRNAs (blue dots)
이 개체들에 대한 유전자 편집 능력을 추가로 평가하기 위해, Guide-it™ Indel Identification Kit를 이용해 목적 서열을 증폭하여 분석한 후 indel을 확인하였다. 두 개체에서 knockout 실험에서 전형적으로 볼 수 있는 indel 패턴의 서열 분석 결과를 확인할 수 있었다 (그림 5).
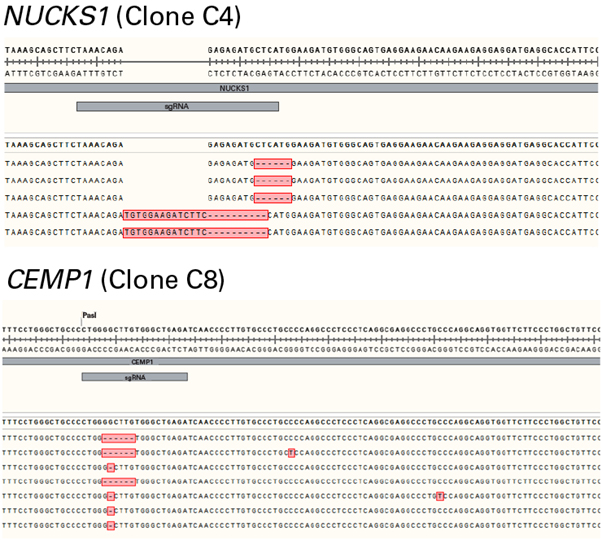
그림 5. Cas9과 sgRNA를 도입한 A375 세포의 두 개체에서 전형적인 indel 패턴 확인
Using the sgRNA library to screen for 6-thioguanine resistance
Purine salvage pathway에 관여하는 유전자를 스크리닝하du 당사의 sgRNA library system의 효율을 확인하였다. Selective agent로 사용된 6-thioguanine (6-TG)는 purine analog로써, hypoxanthine-guanine phosphoribosyl transferase (HPRT) 유전자에 작용하는 세포 내 활성 대사 산물이다. 이는, DNA에 결합하여 DNA mismatch 수복 시스템을 활성화함으로써 세포사멸을 유발한다. 반면, HPRT를 knockout한 세포에서는 6-TG의 처리에도 생존할 수 있게 된다.
Cas9을 발현하는 세포에 낮은 MOI로 sgRNA library virus를 감염시킨 후 hygromycin을 이용해 Cas9과 sgRNA를 모두 발현하는 세포를 선별하여 생존한 세포들을 확대 배양하였다. 이 후, 6-TG를 처리한 군과, 처리하지 않은 reference control, 두 군으로 나누어 배양한 후 gDNA를 추출하여 각각에 대한 NGS 분석을 진행하였다 (그림 6).
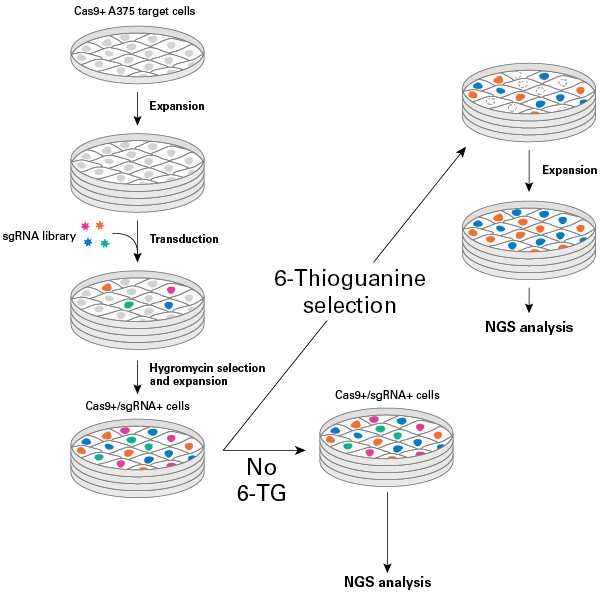
그림 6. 6-thioguanine (6-TG)를 이용한 세포 선별 과정
After transduction of Cas9+ A375 cells with the sgRNA library, hygromycin selection, and expansion, the Cas9+/ sgRNA+ cells were split into two populations. One Cas9+/sgRNA+ cell population was exposed to 6-TG, while the control population was expanded under normal culture conditions without exposure to 6-TG. After expansion of both cell populations, genomic DNA was harvested and prepared for sequencing with the Guide-it CRISPR Genome-Wide sgRNA Library NGS Analysis Kit.
6-TG로 선별한 세포에서 추출한 gDNA로 NGS 분석을 진행했을 때, 대조군에 비해 상당수의 sgRNA가 손실되었으나, HPRT 유전자를 타겟으로 하는 sgRNA는 4종 모두 최대 30,000배 이상 과발현 되었다 (그림 7, Panel B). 또한, 대조군에서는 기존의 plasmid library에서의 sgRNA representation을 보였다 (그림 7, Panel A). Purine 합성 과정에서 HPRT에 의해 코딩된 단백질이 작용할 수 있는 기질을 제공하는 것으로 알려진 NUDT5를 타겟으로 하는 4종의 sgRNA 에서도 유사한 형태의 과발현이 확인되었다 (그림 7, Panel A). Brunello library의 개발자로부터 이와 유사한 형태의 결과가 보고된 바 있다.
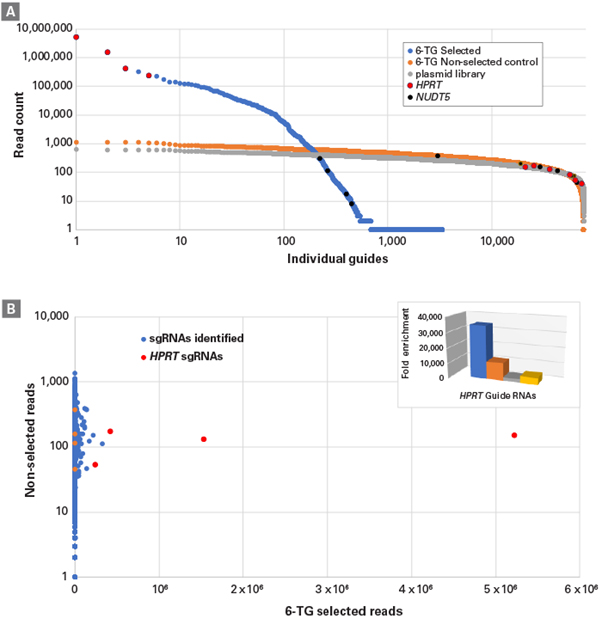
그림 7. 6-TG를 이용한 스크리닝 후 세포로부터 추출한 sgRNA의 판별과 분석
(Panel A) sgRNA representation was compared between the original plasmid library population used to prepare lentivirus, and the resulting transduced cell populations (selected and non-selected). All four sgRNAs targeting HPRT (red dots) and NUDT5 (black dots), respectively, were enriched in the 6-TG-selected population (blue dots). Representation of these sgRNAs in the plasmid library (gray) and transduced, unselected population (orange) is also shown.
(Panel B) After selection, sgRNA representation (blue) was shifted in response to 6-TG. Individual sgRNAs that were enriched are highlighted within this correlation (red and orange dots). The inset shows fold-enrichment of the four sgRNAs targeting the HPRT gene as compared to the plasmid library.
[원문] Performing a phenotypic screen using the Guide-it CRISPR Genome-Wide sgRNA Library System
[참고문헌]
- Chen, B. et al. Dynamic Imaging of Genomic Loci in Living Human Cells by an Optimized CRISPR/Cas System. Cell 155, 1479-1491 (2013).
- Doench, J.G. et al. Optimized sgRNA design to maximize activity and minimize off-target effects of CRISPR-Cas9. Nat. Biotechnol. 34, 184-191 (2016).
- Doench, J.G. Am I ready for CRISPR? A user's guide to genetic screens. Nat. Rev. Genet. 19, 67-80 (2018).
최근, CRISPR/Cas9을 이용해 Whole genome sequencing (WGS)이 가능하다는 것이 확인되면서, 연구자들은 관심이 있는 생물학적 경로에 관련되어 있는 새로운 유전자를 빠르게 식별할 수 있게 되었다. RNAi를 이용한 knockdown과 같은 기존의 방법에 비해 CRISPR/Cas9을 이용한 스크리닝은 보다 확실한 knockout을 이용하여 목적 세포의 표현형을 더 쉽게 검출하고 분석할 수 있다. 이 효과를 극대화하기 위해서는, 사용하기 편리한 시스템을 이용해 인간 세포에 genome-wide phenotypic 스크리닝를 수행할 수 있어야 한다. 다카라바이오에서는 최근의 연구에 기반한 sgRNA library와 Lentivirus system을 접목하여 Guide-it™ CRISPR Genome-Wide sgRNA Library System를 개발하였다.
Library design features
Guide it™ CRISPR/Cas9을 이용한 표현형 스크리닝은 새로운 유전자를 식별하는데 있어, 그 효율을 좌우하는 모든 중요한 프로세스를 제공하고 있다. 제품 내에는 최적화된 sgRNA가 library 형태로 제공될 뿐 아니라 plasmid library 증폭의 최적화, Lentiviral packaging, 목적 세포로의 감염 과정까지 포함하여 구성되어 있다.
본 Library는 19,000개 이상의 단백질 코딩 유전자를 타겟으로, 각 유전자를 타겟으로 하는 sgRNA가 4개씩, 즉 총 76,000개 이상의 sgRNA로 구성되어 있다 (Doench et al. 2016). 더 많은 종류의 sgRNA를 포함한 library를 적용할 수는 있으나, sgRNA의 수가 많아지면 증폭 과정에서 재현성이 감소할 수 있다. 이러한 이유로 multiple plasmid amplification, Lentiviral packaging, target-cell transduction step을 순차적으로 진행하는 다른 library에서는 편향성이 높거나 sgRNA 재현성이 낮은 경우를 확인할 수 있다.
당사의 Library는 Brunello library (Doench et al. 2016)에 기반하여, 다른 library보다 유사한 유전자를 타겟으로 작용하는 등의 off-target effect가 낮고, 목적 유전자에 대한 특이성이 높은 sgRNA를 포함하고 있다.
스크리닝 과정은 어떤 성능의 Library를 선택하는 지가 sgRNA의 재현성을 좌우한다. 이상적인 Library는 각각의 sgRNA가 모두 정확히 동일한 양을 포함하고 있어야 하지만, 이는 sgRNA 합성과 plasmid library 증폭 과정으로 인해 현실적으로 불가능하다. 다카라바이오는 수십년 간 노하우를 바탕으로 cDNA library를 합성하고 증폭하는 과정을 최적화하여, Guide-it™ CRISPR Genome-Wide sgRNA Library System 내 sgRNA library를 굉장히 유사한 정도로 합성하고 증폭할 수 있도록 하였다. 각 lot의 library 내 모든 sgRNA의 90% 이상이 10배 이내의 분포 범위에 위치함을 확인하였다 (그림 1, Panel A).
본 제품은 Lentivirus 생산과 목적 세포로의 감염을 간소화할 수 있는 손쉬운 형태로 library를 제공함으로써, 사용자가 최적화를 진행하지 않고도 형질 도입된 목적 세포에서 sgRNA가 잘 작용할 수 있게 하였다 (그림 1, Panel B).

그림 1. Guide-it™ CRISPR Genome-Wide sgRNA Library System의 sgRNA 재현성 확인
(Panel A) sgRNA representation in the starting plasmid population. The Brunello-based sgRNA library was cloned into the pLVXS-sgRNA-mCherry-hyg Vector and amplified. Representation of the sgRNAs within the plasmid DNA was verified by next-generation sequencing (NGS). Bars represent the number of sgRNAs detected at a given read count within the population. (더 많은 sgRNA representation data를 확인하시려면 클릭하세요)
(Panel B) The correlation between read counts of each integrated sgRNA for the transduced cell population relative to read counts of the corresponding sgRNA for the starting plasmid population. These data show strong Spearman and Pearson correlations, indicating that the Guide-it CRISPR Genome-Wide sgRNA Library System allows for sgRNA representation to be maintained throughout transduction and selection of the target cell population.
Optimized lentiviral sgRNA library vector and sgRNA scaffold design for high editing efficiency
본 제품의 sgRNA library vector로는 pLVXS-sgRNA-mCherry-hyg이 사용 되었고, 성능과 안전성을 모두 고려하여 최적화했다 (그림 2, Panel A). 사용된 Lentiviral vector는 Cas9과 sgRNA를 감염 세포에서 각각 puromycin과 hygromycin로 선별 배양하게 된다. sgRNA library vector는 mCherry 단백질을 형광 리포터로 발현하고 있어, 형광 현미경에서 바로 도입 여부를 판별하고 역가를 확인할 수 있을 뿐 아니라, Cas9을 발현하는 세포주에 sgRNA library를 도입하기 위한 최적의 MOI (multiplicity-of-infection)를 보다 쉽게 결정할 수 있다 (그림 3). MOI는 각 Cas9 발현 세포에서 오직 하나의 sgRNA를 발현할 수 있도록 조절해야 하기에 아주 중요하다.
pLVXS-sgRNA-mCherry-hyg내의 sgRNA scaffold는 유전자 편집 효율을 향상시키기 위해 최적화된 서열로 구성되었으며 (그림 2, Panel B), sgRNA와 Cas9 endonuclease 간의 친화력을 극대화 할 수 있도록 디자인되었다 (Chen et al. 2013). Cas9과 sgRNA vector는 모두 다카라바이오의 Lenti-X™ Packaging Single Shots과 Xfect™ Transfection Reagent와 premix되어 동결 건조 형태로 제공되며, 물을 추가하여 Lenti-X™ 293T Producer Cell Line에 첨가하는 것 만으로 transfection을 위한 준비 과정이 완료된다.

그림 2. Guide-it™ CRISPR sgRNA library에 사용된 vector와 sgRNA scaffold
(Panel A) pLVXS-EF1a-Cas9-PGK-Puro and pLVXS-sgRNA-mCherry-hyg vector maps showing the lentiviral vector backbone. The vectors are self-inactivating for increased safety during production and use. The pLVXS-sgRNA-mCherry-hyg vector contains both mCherry and hygromycin markers expressed from an IRES-linked bicistronic expression cassette.
(Panel B) The sgRNAs, derived from the Brunello library, are expressed from the human U6 promoter and use an optimized scaffold sequence for better Cas9 loading and editing efficiency.

그림 3. mCherry 단백질을 형광 리포터로 이용한 sgRNA library Lentivirus의 도입 효율 확인
sgRNA Library-containing lentivirus was produced following the instructions in the user manual: water was added to a tube of sgRNA Library Transfection Mix, vortexed, incubated for 10 min and then added to the provided Lenti-X 293T cells. Lentivirus was harvested after 48 hours and used to transduce Cas9-expressing A375 cells at varying MOIs. Transduced cells were plated and analyzed for transduction efficiency by fluorescence microscopy after 48 hours.
Results - Editing activity of the Guide-it™ CRISPR Genome-Wide sgRNA Library in randomly selected clones
당사의 sgRNA library system의 편집 효율을 확인하기 위해, 흑색종 세포인 A375 세포에 Cas9과 sgRNA library를 형질 도입하여, 무작위로 20종의 clone을 선택하여 분석하였다. 각 Clone으로부터 sgRNA를 증폭하여 서열을 분석한 후, sgRNA가 타겟으로 하는 유전자에 대한 sgRNA primer를 디자인하였다 (그림 4, Panel A). 편집 효율은 Resolvase에 기반한 Guide-it™ Mutation Detection Kit을 이용하여 확인하였다 (그림 4, Panel B). 모든 개체에서 절단된 것이 확인되었으며, 이를 통해 성공적으로 유전자가 편집 되었음을 확인하였다. 각각의 clone에서 확인된 sgRNA는 실제의 plasmid sgRNA pool과 형질 도입된 세포의 gDNA로 분석한 correlation plot에 mapping 함으로써, 상대적인 read count를 확인하였으며, 각각의 sgRNA는 무작위로 선별되었음에도 plot 분포 내에 위치함이 확인되었다 (그림 4, Panel C).

그림 4. 무작위로 선별된 개체에서 Guide-it™ CRISPR Genome-Wide sgRNA Library System의 편집 효율 확인
(Panel A) After transduction of Cas9+ A375 cells with the sgRNA library and hygromycin selection, twenty clones were selected randomly and expanded for analysis.
(Panel B) Genomic DNA was isolated from each clone and analyzed for activity using the Guide-it Mutation Detection Kit. The results of the resolvase assay for 8 representative clones are shown; NTC: non-transduced cells, (+) PCR products treated with Guide-it Resolvase, (-) untreated PCR products.
(Panel C) The sgRNA sequences from the twenty clones were mapped against the correlation plot of the plasmid library and transduced gDNA. The sgRNAs isolated from the randomly-selected clones are marked with orange dots within the correlation plot of all sgRNAs (blue dots)
이 개체들에 대한 유전자 편집 능력을 추가로 평가하기 위해, Guide-it™ Indel Identification Kit를 이용해 목적 서열을 증폭하여 분석한 후 indel을 확인하였다. 두 개체에서 knockout 실험에서 전형적으로 볼 수 있는 indel 패턴의 서열 분석 결과를 확인할 수 있었다 (그림 5).

그림 5. Cas9과 sgRNA를 도입한 A375 세포의 두 개체에서 전형적인 indel 패턴 확인
Using the sgRNA library to screen for 6-thioguanine resistance
Purine salvage pathway에 관여하는 유전자를 스크리닝하du 당사의 sgRNA library system의 효율을 확인하였다. Selective agent로 사용된 6-thioguanine (6-TG)는 purine analog로써, hypoxanthine-guanine phosphoribosyl transferase (HPRT) 유전자에 작용하는 세포 내 활성 대사 산물이다. 이는, DNA에 결합하여 DNA mismatch 수복 시스템을 활성화함으로써 세포사멸을 유발한다. 반면, HPRT를 knockout한 세포에서는 6-TG의 처리에도 생존할 수 있게 된다.
Cas9을 발현하는 세포에 낮은 MOI로 sgRNA library virus를 감염시킨 후 hygromycin을 이용해 Cas9과 sgRNA를 모두 발현하는 세포를 선별하여 생존한 세포들을 확대 배양하였다. 이 후, 6-TG를 처리한 군과, 처리하지 않은 reference control, 두 군으로 나누어 배양한 후 gDNA를 추출하여 각각에 대한 NGS 분석을 진행하였다 (그림 6).

그림 6. 6-thioguanine (6-TG)를 이용한 세포 선별 과정
After transduction of Cas9+ A375 cells with the sgRNA library, hygromycin selection, and expansion, the Cas9+/ sgRNA+ cells were split into two populations. One Cas9+/sgRNA+ cell population was exposed to 6-TG, while the control population was expanded under normal culture conditions without exposure to 6-TG. After expansion of both cell populations, genomic DNA was harvested and prepared for sequencing with the Guide-it CRISPR Genome-Wide sgRNA Library NGS Analysis Kit.
6-TG로 선별한 세포에서 추출한 gDNA로 NGS 분석을 진행했을 때, 대조군에 비해 상당수의 sgRNA가 손실되었으나, HPRT 유전자를 타겟으로 하는 sgRNA는 4종 모두 최대 30,000배 이상 과발현 되었다 (그림 7, Panel B). 또한, 대조군에서는 기존의 plasmid library에서의 sgRNA representation을 보였다 (그림 7, Panel A). Purine 합성 과정에서 HPRT에 의해 코딩된 단백질이 작용할 수 있는 기질을 제공하는 것으로 알려진 NUDT5를 타겟으로 하는 4종의 sgRNA 에서도 유사한 형태의 과발현이 확인되었다 (그림 7, Panel A). Brunello library의 개발자로부터 이와 유사한 형태의 결과가 보고된 바 있다.

그림 7. 6-TG를 이용한 스크리닝 후 세포로부터 추출한 sgRNA의 판별과 분석
(Panel A) sgRNA representation was compared between the original plasmid library population used to prepare lentivirus, and the resulting transduced cell populations (selected and non-selected). All four sgRNAs targeting HPRT (red dots) and NUDT5 (black dots), respectively, were enriched in the 6-TG-selected population (blue dots). Representation of these sgRNAs in the plasmid library (gray) and transduced, unselected population (orange) is also shown.
(Panel B) After selection, sgRNA representation (blue) was shifted in response to 6-TG. Individual sgRNAs that were enriched are highlighted within this correlation (red and orange dots). The inset shows fold-enrichment of the four sgRNAs targeting the HPRT gene as compared to the plasmid library.
Code |
제품명 |
용량 |
632646 |
5 회 |
|
632647 |
10 회 |
|
632651 |
20 회 |
|
631448 |
25 회 |
|
631444 |
10 회 |
[원문] Performing a phenotypic screen using the Guide-it CRISPR Genome-Wide sgRNA Library System
[참고문헌]
- Chen, B. et al. Dynamic Imaging of Genomic Loci in Living Human Cells by an Optimized CRISPR/Cas System. Cell 155, 1479-1491 (2013).
- Doench, J.G. et al. Optimized sgRNA design to maximize activity and minimize off-target effects of CRISPR-Cas9. Nat. Biotechnol. 34, 184-191 (2016).
- Doench, J.G. Am I ready for CRISPR? A user's guide to genetic screens. Nat. Rev. Genet. 19, 67-80 (2018).