- 고객지원
- 업무안내│제품문의
- 전화번호│02-2081-2510
- Email │support@takara.co.kr
- 대전지사
- 업무안내│대전/충청지역 주문, 제품문의
- 전화번호│042-828-6525
- Email │tkbd@takara.co.kr
- 업무시간안내
- [ 평 일 ] 09 : 00 ~ 18 : 00 │ [ 점심시간 ] 12 : 00 ~ 13 : 00
- 토·일요일, 공휴일은 휴무입니다.
[Cancer Research] Cancer single cell analysis


Accurate detection of SNVs and CNVs from five-cell inputs in a single, low-pass sequencing run
적은 양의 DNA 샘플로부터 copy-number variants (CNVs)와 single-nucleotide variants (SNVs)를 정확하게 검출하는 것은 종양 연구에서 유용하게 활용될 수 있지만 각각의 샘플에 대한 library를 제작하는 과정이 필요하기 때문에 많은 비용이 소요된다.
CNV와 SNV를 검출도 높은 depth로 whole-genome sequencing (WGS)를 진행하는 것이 좋으나, 이 또한 비용과 노력이 많이 요구된다. 빈도가 적은 SNV를 확인할 수 있을 정도의 depth로 분석하기 해서는 targeted sequencing을 이용하는 것이 일반적이지만, 이는 한정된 게놈 영역에서만 분석할 수 있다. 또한, CNV 분석을 위해서는 library 제작과 sequencing 과정을 별도로 진행해야 한다.
다카라바이오는 Amplicon-based enrichment를 활용하여 고품질의 library를 제작하여, 샘플 당 백만개의 reads만을 읽는 shallow sequencing을 통해 targeted SNV와 genome-wide CNV를 검출할 수 있었다. 금상첨화로, library는 하루 안에 제작 가능하며, 실험 시간과 노동력을 크게 절감할 수 있다. 이는 CNV, SNV 검출을 위해 별도의 library 제작과 deep sequencing이 요구되던 기존의 whole-genome 분석법과 대조적이다.
이 기술을 증명하기 위해, 다카라바이오의 PicoPLEX® Gold Single Cell DNA-Seq Kit (PicoPLEX® Gold)와 Illumina®사의 AmpliSeq을 이용하여 5개의 세포에서 50만개의 75 bp paired-ended reads로 shallow sequencing하였다. 이 실험 과정은 하루 내 완료 되었으며, 시중에 판매되는 어떤 기술을 이용하였을 때 보다 빠르고 경제적이었다 (그림 1).
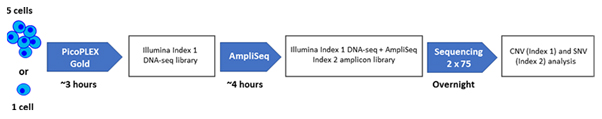
그림 1. PicoPLEX Gold와 AmpliSeq Hot-Spot Cancer Panel을 이용한 SNV, CNV의 분석 과정
PicoPLEX Gold + AmpliSeq = SNV and CNV detection in a single sequencing run
GM12878 세포 5개씩 2개의 샘플을 이용하여 AmpliSeq for Illumina® Cancer Hotspot Panel v2와 PicoPLEX Gold로 enrichment하여 DNA-seq library를 제작하였으며, 두 개의 샘플에서 제작한 library는 high amplicon-coverage 재현성과 100%의 변이 검출, high bin-level genome coverage correlation, 염색체 간 low noise levels을 보임을 확인하였다.
Uniform and reproducible library size distribution and amplicon coverage
PicoPLEX® gold와 AmpliSeq panel로 제작된 NGS library는 bioanalyzer를 통해 high fragment-size reproducibility를 보이고 있음을 확인하였다 (그림 2). Whole-genome에 대한 DNA-seq library는 기본적으로 amplicon enrichment 과정에 의해 수정되거나 손상되지 않았으며, 207 amplicon 중 206개는 최소 100X의 depth에서 균일하고 재현성 높게 관찰되었다. 이를 통해 두 library에서 모두 high amplicon-coverage의 재현성을 확인할 수 있었다.
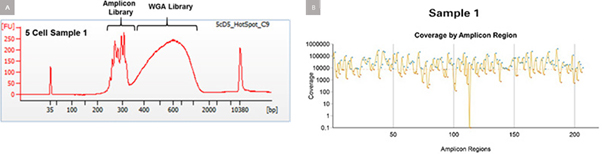
그림 2. Bioanalyzer traces and amplicon coverage depth of representative library.
(Panel A) Bioanalyzer trace shows the expected size separation between the whole-genome NGS library and the amplicon library.
(Panel B) Coverage plot shows the read depth coverage of each individual amplicon plotted on a log10 scale. For this sample, amplicon mean coverage was 38,037.5 with 67.63% uniformity.
Variant detection analysis
GM12878 세포의 reference genome은 본 연구에 사용된 22 kb의 amplicon panel에서 3개의 homozygous한 변이와 8개의 heterozygous한 변이를 포함하고 있다. PicoPLEX® gold와 AmpliSeq panel으로 분석한 두 개의 샘플에서 11종의 모든 변이를 보였다. 두 실험군에서 모두 Allele dropout이 없고, heterozygous하 변이는 예상했던 수치인 50%에 가까웠으며, 위양성을 띄는 1개의 amplicon을 확인했다. Homozygous한 변이에 대한 alternate allele의 검출 확률은 99~100%인 반면, 8개의 heterozygous한 변이의 검출 확률은 33~86%였다 (Allele depth 30X 이상) (표 1).
*For the GM12878 cell line, there were eleven variants contained in the Hot-Spot Cancer Panel.
**Filters: Minor allele frequency >0.3, Variant depth >10
***Total panel size=22 kb
표 1. 5개의 세포를 PicoPLEX® Gold와 AmpliSeq panel에서 각각 분석했을 때 검출된 변이
The data shows detection of three homozygous and eight heterozygous variants found in the GM12878 genome along the 22-kb amplicon panel, and concordance with the bulk DNA genotype. The false positive percentage was calculated as discordance with the reference at any position of the 22-kb amplicon panel.
[원문] Accurate detection of SNVs and CNVs from five-cell inputs in a single, low-pass sequencing run
적은 양의 DNA 샘플로부터 copy-number variants (CNVs)와 single-nucleotide variants (SNVs)를 정확하게 검출하는 것은 종양 연구에서 유용하게 활용될 수 있지만 각각의 샘플에 대한 library를 제작하는 과정이 필요하기 때문에 많은 비용이 소요된다.
CNV와 SNV를 검출도 높은 depth로 whole-genome sequencing (WGS)를 진행하는 것이 좋으나, 이 또한 비용과 노력이 많이 요구된다. 빈도가 적은 SNV를 확인할 수 있을 정도의 depth로 분석하기 해서는 targeted sequencing을 이용하는 것이 일반적이지만, 이는 한정된 게놈 영역에서만 분석할 수 있다. 또한, CNV 분석을 위해서는 library 제작과 sequencing 과정을 별도로 진행해야 한다.
다카라바이오는 Amplicon-based enrichment를 활용하여 고품질의 library를 제작하여, 샘플 당 백만개의 reads만을 읽는 shallow sequencing을 통해 targeted SNV와 genome-wide CNV를 검출할 수 있었다. 금상첨화로, library는 하루 안에 제작 가능하며, 실험 시간과 노동력을 크게 절감할 수 있다. 이는 CNV, SNV 검출을 위해 별도의 library 제작과 deep sequencing이 요구되던 기존의 whole-genome 분석법과 대조적이다.
이 기술을 증명하기 위해, 다카라바이오의 PicoPLEX® Gold Single Cell DNA-Seq Kit (PicoPLEX® Gold)와 Illumina®사의 AmpliSeq을 이용하여 5개의 세포에서 50만개의 75 bp paired-ended reads로 shallow sequencing하였다. 이 실험 과정은 하루 내 완료 되었으며, 시중에 판매되는 어떤 기술을 이용하였을 때 보다 빠르고 경제적이었다 (그림 1).

그림 1. PicoPLEX Gold와 AmpliSeq Hot-Spot Cancer Panel을 이용한 SNV, CNV의 분석 과정
PicoPLEX Gold + AmpliSeq = SNV and CNV detection in a single sequencing run
GM12878 세포 5개씩 2개의 샘플을 이용하여 AmpliSeq for Illumina® Cancer Hotspot Panel v2와 PicoPLEX Gold로 enrichment하여 DNA-seq library를 제작하였으며, 두 개의 샘플에서 제작한 library는 high amplicon-coverage 재현성과 100%의 변이 검출, high bin-level genome coverage correlation, 염색체 간 low noise levels을 보임을 확인하였다.
Uniform and reproducible library size distribution and amplicon coverage
PicoPLEX® gold와 AmpliSeq panel로 제작된 NGS library는 bioanalyzer를 통해 high fragment-size reproducibility를 보이고 있음을 확인하였다 (그림 2). Whole-genome에 대한 DNA-seq library는 기본적으로 amplicon enrichment 과정에 의해 수정되거나 손상되지 않았으며, 207 amplicon 중 206개는 최소 100X의 depth에서 균일하고 재현성 높게 관찰되었다. 이를 통해 두 library에서 모두 high amplicon-coverage의 재현성을 확인할 수 있었다.

그림 2. Bioanalyzer traces and amplicon coverage depth of representative library.
(Panel A) Bioanalyzer trace shows the expected size separation between the whole-genome NGS library and the amplicon library.
(Panel B) Coverage plot shows the read depth coverage of each individual amplicon plotted on a log10 scale. For this sample, amplicon mean coverage was 38,037.5 with 67.63% uniformity.
Variant detection analysis
GM12878 세포의 reference genome은 본 연구에 사용된 22 kb의 amplicon panel에서 3개의 homozygous한 변이와 8개의 heterozygous한 변이를 포함하고 있다. PicoPLEX® gold와 AmpliSeq panel으로 분석한 두 개의 샘플에서 11종의 모든 변이를 보였다. 두 실험군에서 모두 Allele dropout이 없고, heterozygous하 변이는 예상했던 수치인 50%에 가까웠으며, 위양성을 띄는 1개의 amplicon을 확인했다. Homozygous한 변이에 대한 alternate allele의 검출 확률은 99~100%인 반면, 8개의 heterozygous한 변이의 검출 확률은 33~86%였다 (Allele depth 30X 이상) (표 1).
True positives* |
% True positives |
Allele dropouts** |
False positives*** |
% False positives |
|
Reference |
11 |
100 |
0 |
0 |
0 |
Sample 1 |
11 |
100 |
0 |
1 |
4.5E-3 |
Sample 2 |
11 |
100 |
0 |
1 |
4.5E-3 |
*For the GM12878 cell line, there were eleven variants contained in the Hot-Spot Cancer Panel.
**Filters: Minor allele frequency >0.3, Variant depth >10
***Total panel size=22 kb
표 1. 5개의 세포를 PicoPLEX® Gold와 AmpliSeq panel에서 각각 분석했을 때 검출된 변이
The data shows detection of three homozygous and eight heterozygous variants found in the GM12878 genome along the 22-kb amplicon panel, and concordance with the bulk DNA genotype. The false positive percentage was calculated as discordance with the reference at any position of the 22-kb amplicon panel.
Code |
제품명 |
용량 |
R300669 |
24 회 |
[원문] Accurate detection of SNVs and CNVs from five-cell inputs in a single, low-pass sequencing run