- 고객지원
- 업무안내│제품문의
- 전화번호│02-2081-2510
- Email │support@takara.co.kr
- 업무시간안내
- [ 평 일 ] 09 : 00 ~ 18 : 00 │ [ 점심시간 ] 12 : 00 ~ 13 : 00
- 토·일요일, 공휴일은 휴무입니다.
[Cancer Research] Cancer Genomics & Epigenomics

Cancer genomics and epigenomics
Mutation이나 copy number variation, 구조적 변이 등과 같은 게놈 변이, 후성 유전학적 환경으로 유발되는 조절 장애 (i.e., DNA methylation status) 등은 암에서 보이는 전형적인 특징으로 알려져 있다. 암 연구자들은 Whole genome sequencing (WGS)와 targeted sequencing를 통해 주로 체세포 변이나 CNV를 검출함으로써, 게놈 변이를 포괄적으로 이해할 수 있게 되었다. Exome이나 panel sequencing과 같이 특정 유전자나 특정 부위를 표적 분석함으로써, WGS에서 확인한 변이의 검출 민감도 보다 더 높은 depth로 분석할 수 있다. 반면에 후성 유전학적으로 접근하면, 암 특이적 DNA와 결합하는 단백질이나 히스톤 변형, 암세포 내의 DNA methylation을 효과적으로 규명해낼 수 있다. 유전적 변이를 동반하는 이러한 후성 유전학적 변화가 종양 생성과 진행, 치료 내성 등에 어떻게 관여하는지 이해하는 것은 종양 치료법의 발전을 위해 매우 중요하다.
Highlighted products
다카라바이오는 체세포 변이와 CNV 검출을 위한 WGS와 targeted sequencing 과정을 보다 빠르게 진행할 수 있도록 ThruPLEX®를 포함한 다양한 혁신 기술을 제공하고 있다. 다카라바이오의 ThruPLEX® 제품은 사용 시에 발생할 수 있는 error나 샘플 손실, 오염 물질 혼입을 방지하기 위해 single tube 내 3-step 과정으로 2시간 내 완료할 수 있다 (그림 1).
ThruPLEX® DNA-Seq Kit를 이용하면 FFPE 샘플이나 ChIP DNA를 포함한 picogram 수준의 DNA로부터 NGS 라이브러리를 구축할 수 있다. ThruPLEX® Tag-seq Kit은 증폭 전 DNA 조각 각각을 tagging 하는 1,600만개의 unique sequence를 포함하고 있어, 라이브러리의 준비나 target enrichment, 데이터 분석 과정에서 저빈도 대립유전자를 검출하거나 각각의 단편을 확인하는 데 효과적이다.
ThruPLEX® 기술은 Agilent SureSelect, Roche Nimblegen SeqCap EZ나 IDT xGen Lockdown probes와 같은 target enrichment 플랫폼에도 적용하여 사용할 수 있다. 또한, 다양한 종류의 암에 대한 WGS, targeted sequencing, CNV 분석 및 ChIP-seq 연구에서 성공적으로 활용되고 있음을 다양한 참고 문헌에서 확인할 수 있다.
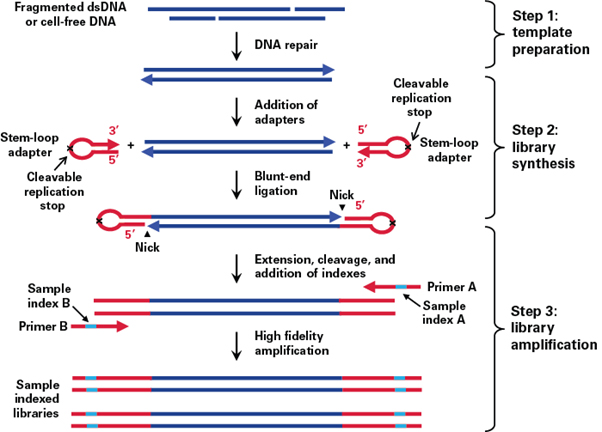
그림 1. ThruPLEX® 기술을 이용한 실험 과정
[원문] Cancer genomics and epigenomics
[참고문헌] References and publications citing the use of SMARTer ThruPLEX technology for whole genome sequencing, targeted sequencing, CNV analysis, and ChIP-seq studies in various types of cancer include the following:
- Cato, L. et al. Development of Bag-1L as a therapeutic target in androgen receptor-dependent prostate cancer. eLife 6, e27159 (2017).
- Jeselsohn, R. et al. Allele-specific chromatin recruitment and therapeutic vulnerabilities of ESR1 activating mutations. Cancer Cell 33, 173-186 (2018).
- Jin, X. et al. Targeting glioma stem cells through combined BMI1 and EZH2 inhibition. Nature Med. 23, 1352-1361 (2017).
- Klevebring, D. et al. Evaluation of exome sequencing to estimate tumor burden in plasma. PLoS One 18, e104417 (2014).
- Markus, H. et al. Evaluation of pre-analytical factors affecting plasma DNA analysis. Sci. Rep. 8, 7375 (2018).
- McNair, C. et al. Differential impact of RB status on E2F1 reprogramming in human cancer. J. Clin. Invest. 128, 341-358 (2018).
- Murtaza, M. et al. Non-invasive analysis of acquired resistance to cancer therapy by sequencing of plasma DNA. Nature 497, 108-112 (2013).
- Patel, K. M. et al. Association of plasma and urinary mutant DNA with clinical outcomes in muscle-invasive bladder cancer. Sci. Rep. 7, 5554 (2017).
- Wang, X. et al. Purine synthesis promotes maintenance of brain tumor-initiating cells in glioma. Nature Neurosci. 20, 661-673 (2017).
- Weiss, G. J. et al. Tumor cell-free DNA copy number instability predicts therapeutic response to immunotherapy. Clin. Cancer Res. 23, 5074-5081 (2017).
Mutation이나 copy number variation, 구조적 변이 등과 같은 게놈 변이, 후성 유전학적 환경으로 유발되는 조절 장애 (i.e., DNA methylation status) 등은 암에서 보이는 전형적인 특징으로 알려져 있다. 암 연구자들은 Whole genome sequencing (WGS)와 targeted sequencing를 통해 주로 체세포 변이나 CNV를 검출함으로써, 게놈 변이를 포괄적으로 이해할 수 있게 되었다. Exome이나 panel sequencing과 같이 특정 유전자나 특정 부위를 표적 분석함으로써, WGS에서 확인한 변이의 검출 민감도 보다 더 높은 depth로 분석할 수 있다. 반면에 후성 유전학적으로 접근하면, 암 특이적 DNA와 결합하는 단백질이나 히스톤 변형, 암세포 내의 DNA methylation을 효과적으로 규명해낼 수 있다. 유전적 변이를 동반하는 이러한 후성 유전학적 변화가 종양 생성과 진행, 치료 내성 등에 어떻게 관여하는지 이해하는 것은 종양 치료법의 발전을 위해 매우 중요하다.
Highlighted products
다카라바이오는 체세포 변이와 CNV 검출을 위한 WGS와 targeted sequencing 과정을 보다 빠르게 진행할 수 있도록 ThruPLEX®를 포함한 다양한 혁신 기술을 제공하고 있다. 다카라바이오의 ThruPLEX® 제품은 사용 시에 발생할 수 있는 error나 샘플 손실, 오염 물질 혼입을 방지하기 위해 single tube 내 3-step 과정으로 2시간 내 완료할 수 있다 (그림 1).
ThruPLEX® DNA-Seq Kit를 이용하면 FFPE 샘플이나 ChIP DNA를 포함한 picogram 수준의 DNA로부터 NGS 라이브러리를 구축할 수 있다. ThruPLEX® Tag-seq Kit은 증폭 전 DNA 조각 각각을 tagging 하는 1,600만개의 unique sequence를 포함하고 있어, 라이브러리의 준비나 target enrichment, 데이터 분석 과정에서 저빈도 대립유전자를 검출하거나 각각의 단편을 확인하는 데 효과적이다.
ThruPLEX® 기술은 Agilent SureSelect, Roche Nimblegen SeqCap EZ나 IDT xGen Lockdown probes와 같은 target enrichment 플랫폼에도 적용하여 사용할 수 있다. 또한, 다양한 종류의 암에 대한 WGS, targeted sequencing, CNV 분석 및 ChIP-seq 연구에서 성공적으로 활용되고 있음을 다양한 참고 문헌에서 확인할 수 있다.

그림 1. ThruPLEX® 기술을 이용한 실험 과정
Code |
제품명 |
용량 |
R400674 |
24회 |
|
R400741 |
24회 |
|
R400584 |
24회 |
|
R400742 |
24회 |
[원문] Cancer genomics and epigenomics
[참고문헌] References and publications citing the use of SMARTer ThruPLEX technology for whole genome sequencing, targeted sequencing, CNV analysis, and ChIP-seq studies in various types of cancer include the following:
- Cato, L. et al. Development of Bag-1L as a therapeutic target in androgen receptor-dependent prostate cancer. eLife 6, e27159 (2017).
- Jeselsohn, R. et al. Allele-specific chromatin recruitment and therapeutic vulnerabilities of ESR1 activating mutations. Cancer Cell 33, 173-186 (2018).
- Jin, X. et al. Targeting glioma stem cells through combined BMI1 and EZH2 inhibition. Nature Med. 23, 1352-1361 (2017).
- Klevebring, D. et al. Evaluation of exome sequencing to estimate tumor burden in plasma. PLoS One 18, e104417 (2014).
- Markus, H. et al. Evaluation of pre-analytical factors affecting plasma DNA analysis. Sci. Rep. 8, 7375 (2018).
- McNair, C. et al. Differential impact of RB status on E2F1 reprogramming in human cancer. J. Clin. Invest. 128, 341-358 (2018).
- Murtaza, M. et al. Non-invasive analysis of acquired resistance to cancer therapy by sequencing of plasma DNA. Nature 497, 108-112 (2013).
- Patel, K. M. et al. Association of plasma and urinary mutant DNA with clinical outcomes in muscle-invasive bladder cancer. Sci. Rep. 7, 5554 (2017).
- Wang, X. et al. Purine synthesis promotes maintenance of brain tumor-initiating cells in glioma. Nature Neurosci. 20, 661-673 (2017).
- Weiss, G. J. et al. Tumor cell-free DNA copy number instability predicts therapeutic response to immunotherapy. Clin. Cancer Res. 23, 5074-5081 (2017).