- 고객지원
- 업무안내│제품문의
- 전화번호│02-2081-2510
- Email │support@takara.co.kr
- 대전지사
- 업무안내│대전/충청지역 주문, 제품문의
- 전화번호│042-828-6525
- Email │tkbd@takara.co.kr
- 업무시간안내
- [ 평 일 ] 09 : 00 ~ 18 : 00 │ [ 점심시간 ] 12 : 00 ~ 13 : 00
- 토·일요일, 공휴일은 휴무입니다.
[Cancer Research] Cancer Genomics & Epigenomics

Bringing epigenomic profiling to the single-cell biology stage
간혹 연구자들은 기발하고 혁신적인 연구를 실행할 방법을 찾기 힘들 때가 있다. 이처럼 이전에는 불가능했던 새로운 연구분야를 위해서는 이를 적용할 수 있는 도구가 뒷받침되어야 한다.
ICELL8® Single-Cell System은 이러한 상황에도 적용할 수 있도록 open platform으로 설계되었으며, 실제로 많은 연구자들이 이를 활용하고 있다. Fred Hutchinson Cancer Research 소속의 Steven Henikoff 박사 연구그룹을 예로 들어볼 수 있으며, CUT&Tag (Cleavage Under Targets and Tagmentation)을 이용한 새로운 후성 유전학 연구 기법을 개발하여 single cell에서 높은 수준의 chromatin mapping을 가능케 했다 (Kaya-Okur et al. 2019).
Going beyond ChIP-seq
후성 유전학은 동일한 Genotype에서 DNA 구조와 히스톤의 변형, DNA methylation으로 인해 발생하는 유전자 발현과 표현형 변화를 이해하는 학문이다 (Deichmann 2016). 이러한 후성 유전학적 분석을 위해 ChIP-seq이 많은 역할을 했으나, 분석을 위한 샘플 양이 충분하지 않거나 높은 백그라운드로 인해 분석 결과가 좋지 않는 등의 한계가 있었다.
적은 수의 세포를 분석하기 위해 낮은 백그라운드를 가지는 실험 방법을 개발하는 것은 매우 중요했다. 2017년 Skene 박사와 Henikoff 박사는 Transcription-factor-DNA (TF-DNA) complex의 특정 DNA 단편 양 말단에 존재하면서 항체와 결합하는 특징을 이용하여, protein A-monococcal nuclease (protein A-MNase)와 결합시켜 ChIP-seq의 한계점을 극복하는 CUT&RUN을 개발해냈다.
Getting to the single-cell level
CUT&RUN 방법은 자동화가 가능하고, 적은 샘플 양으로도 분석이 가능함에도 불구하고, Dr. Henikoff 박사와 연구그룹은 이를 좀 더 개선하고자 했다. 특히 TF-DNA complex를 이용해 library를 제작할 때, DNA 말단 제거와 adapter ligation 단계를 생략하고자 했다. 본 연구 그룹은 기존의 Mnase가 결합된 protein A를 hyperactive Tn5 transposase (pA-Tn5)가 결합된 protein A로 대체하여 CUT&Tag 방법을 개발하였다. pA-Tn5가 활성화 되게 되면, 사전에 로딩된 sequencing adapter가 TF-DNA 양 말단과 부착됨으로써 PCR 증폭을 위한 산물이 생성되며, ChIP-seq 실험과 비교했을 때 분석 범위가 20배 가량 향상되었다.
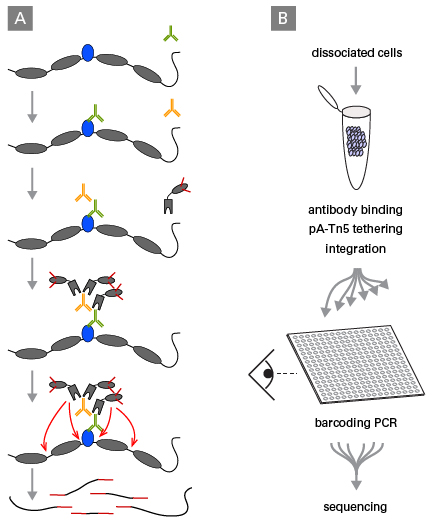
그림 1. ICELL8 Single-Cell System을 이용한 CUT&Tag 과정
(Panel A) Schematic depicting the CUT&Tag process. The target protein (blue) is bound by the primary antibody (green), which is then bound by a secondary antibody (orange). An adapter (red)-loaded Tn5 transposase conjugated to protein A (grey) then binds the secondary antibody, tethering it and promoting the incorporation of adapters at the target site. Fragments are then enriched by PCR.
(Panel B) Workflow of single-cell CUT&Tag as performed on our ICELL8 Single-Cell System. (Figure by Kaya-Okur et al. 2019, used under CC-BY-NC-ND 4.0)
Kaya-Okur et al. 논문에서는 CUT&Tag 방법이 single cell에도 적용 가능한지를 확인하였다 (scCUT&Tag). 해당 프로토콜은 세포의 Permeabilization 과정이 요구되기에, 손상되기 쉬운 세포를 single cell로 분리할 수 있는 시스템을 필요로 했다. 다카라바이오의 ICELL8 자동화 플랫폼을 이용하면 이 permeabilized된 세포를 5,184개의 well을 가지는 chip에 분주하고 tagmentation을 진행할 수 있다. 게다가, 시스템의 이미지 통합 기능을 통해 Well 내의 세포를 시각적으로 확인할 수 있어, 하나의 세포만을 가지는 well에 시약을 선택적으로 적용하여 PCR enrichment 과정을 진행할 수 있다. 이후 연구자들은 ICELL8® System의 스크립트를 이용해서 각 72개의 i5와 i7 index를 분주하여 각각의 세포가 고유한 index를 갖도록 한 후, 이를 추적 분석하였다. 본 논문의 저자들은 이 과정에서 scCUT&Tag 방법을 이용한 실험 결과가 chromatin profiling 결과와 잘 일치함을 확인하였다 (논문 내 그림 6).
[원문] Bringing epigenomic profiling to the single-cell biology stage
[참고문헌]
- Deichmann, U. Epigenetics: The origins and evolution of a fashionable topic. Dev. Biol. 416, 249-254 (2016).
- Kaya-Okur, H. S. et al. CUT&Tag for efficient epigenomic profiling of small samples and single cells. bioRxiv 568915 (2019).
- Skene, P. J. & Henikoff, S. An efficient targeted nuclease strategy for high-resolution mapping of DNA binding sites. Elife 6, (2017).
간혹 연구자들은 기발하고 혁신적인 연구를 실행할 방법을 찾기 힘들 때가 있다. 이처럼 이전에는 불가능했던 새로운 연구분야를 위해서는 이를 적용할 수 있는 도구가 뒷받침되어야 한다.
ICELL8® Single-Cell System은 이러한 상황에도 적용할 수 있도록 open platform으로 설계되었으며, 실제로 많은 연구자들이 이를 활용하고 있다. Fred Hutchinson Cancer Research 소속의 Steven Henikoff 박사 연구그룹을 예로 들어볼 수 있으며, CUT&Tag (Cleavage Under Targets and Tagmentation)을 이용한 새로운 후성 유전학 연구 기법을 개발하여 single cell에서 높은 수준의 chromatin mapping을 가능케 했다 (Kaya-Okur et al. 2019).
Going beyond ChIP-seq
후성 유전학은 동일한 Genotype에서 DNA 구조와 히스톤의 변형, DNA methylation으로 인해 발생하는 유전자 발현과 표현형 변화를 이해하는 학문이다 (Deichmann 2016). 이러한 후성 유전학적 분석을 위해 ChIP-seq이 많은 역할을 했으나, 분석을 위한 샘플 양이 충분하지 않거나 높은 백그라운드로 인해 분석 결과가 좋지 않는 등의 한계가 있었다.
적은 수의 세포를 분석하기 위해 낮은 백그라운드를 가지는 실험 방법을 개발하는 것은 매우 중요했다. 2017년 Skene 박사와 Henikoff 박사는 Transcription-factor-DNA (TF-DNA) complex의 특정 DNA 단편 양 말단에 존재하면서 항체와 결합하는 특징을 이용하여, protein A-monococcal nuclease (protein A-MNase)와 결합시켜 ChIP-seq의 한계점을 극복하는 CUT&RUN을 개발해냈다.
Getting to the single-cell level
CUT&RUN 방법은 자동화가 가능하고, 적은 샘플 양으로도 분석이 가능함에도 불구하고, Dr. Henikoff 박사와 연구그룹은 이를 좀 더 개선하고자 했다. 특히 TF-DNA complex를 이용해 library를 제작할 때, DNA 말단 제거와 adapter ligation 단계를 생략하고자 했다. 본 연구 그룹은 기존의 Mnase가 결합된 protein A를 hyperactive Tn5 transposase (pA-Tn5)가 결합된 protein A로 대체하여 CUT&Tag 방법을 개발하였다. pA-Tn5가 활성화 되게 되면, 사전에 로딩된 sequencing adapter가 TF-DNA 양 말단과 부착됨으로써 PCR 증폭을 위한 산물이 생성되며, ChIP-seq 실험과 비교했을 때 분석 범위가 20배 가량 향상되었다.

그림 1. ICELL8 Single-Cell System을 이용한 CUT&Tag 과정
(Panel A) Schematic depicting the CUT&Tag process. The target protein (blue) is bound by the primary antibody (green), which is then bound by a secondary antibody (orange). An adapter (red)-loaded Tn5 transposase conjugated to protein A (grey) then binds the secondary antibody, tethering it and promoting the incorporation of adapters at the target site. Fragments are then enriched by PCR.
(Panel B) Workflow of single-cell CUT&Tag as performed on our ICELL8 Single-Cell System. (Figure by Kaya-Okur et al. 2019, used under CC-BY-NC-ND 4.0)
Kaya-Okur et al. 논문에서는 CUT&Tag 방법이 single cell에도 적용 가능한지를 확인하였다 (scCUT&Tag). 해당 프로토콜은 세포의 Permeabilization 과정이 요구되기에, 손상되기 쉬운 세포를 single cell로 분리할 수 있는 시스템을 필요로 했다. 다카라바이오의 ICELL8 자동화 플랫폼을 이용하면 이 permeabilized된 세포를 5,184개의 well을 가지는 chip에 분주하고 tagmentation을 진행할 수 있다. 게다가, 시스템의 이미지 통합 기능을 통해 Well 내의 세포를 시각적으로 확인할 수 있어, 하나의 세포만을 가지는 well에 시약을 선택적으로 적용하여 PCR enrichment 과정을 진행할 수 있다. 이후 연구자들은 ICELL8® System의 스크립트를 이용해서 각 72개의 i5와 i7 index를 분주하여 각각의 세포가 고유한 index를 갖도록 한 후, 이를 추적 분석하였다. 본 논문의 저자들은 이 과정에서 scCUT&Tag 방법을 이용한 실험 결과가 chromatin profiling 결과와 잘 일치함을 확인하였다 (논문 내 그림 6).
Code |
제품명 |
용량 |
640189 |
1 System |
[원문] Bringing epigenomic profiling to the single-cell biology stage
[참고문헌]
- Deichmann, U. Epigenetics: The origins and evolution of a fashionable topic. Dev. Biol. 416, 249-254 (2016).
- Kaya-Okur, H. S. et al. CUT&Tag for efficient epigenomic profiling of small samples and single cells. bioRxiv 568915 (2019).
- Skene, P. J. & Henikoff, S. An efficient targeted nuclease strategy for high-resolution mapping of DNA binding sites. Elife 6, (2017).