- 고객지원
- 업무안내│제품문의
- 전화번호│02-2081-2510
- Email │support@takara.co.kr
- 대전지사
- 업무안내│대전/충청지역 주문, 제품문의
- 전화번호│042-828-6525
- Email │tkbd@takara.co.kr
- 업무시간안내
- [ 평 일 ] 09 : 00 ~ 18 : 00 │ [ 점심시간 ] 12 : 00 ~ 13 : 00
- 토·일요일, 공휴일은 휴무입니다.
[Cancer Research] HLA typing


Takara LA Taq®
1) Lan, James H., et al. "Impact of three Illumina library construction methods on GC bias and HLA genotype calling." Human Immunology 76.2-3 (2015): 166-175. 내 발췌
NGS는 구분이 어려운 대립 유전자를 확인하고 높은 정확도로 HLA typing이 가능한 것으로 점차 인식되고 있다. HLA 유전자는 다형성의 특징을 가지고 있어, NGS를 통해 이를 분석하는 경우 종종 균일하지 않은 reads가 분포함으로써 변이 검출의 신뢰도를 낮출 수 있다. 최근, Coverage bias를 유발하는 요인이 library 제작 과정에서 유발된다는 사실이 확인된 바 있다. HLA genotyping 과정에서의 영향을 확인하기 위해, 본 논문의 저자는 먼저, 12개 샘플을 대상으로 Takara LA Taq® 을 이용해 long-range PCR을 진행하였다. 각 샘플에서 HLA-A, -B, -C, -DRB1, -DQB1에 대한 유전자를 증폭한 뒤, TruSeq Nano, Nextera, Nextera XT, 총 세개의 Illumina® library 제작 제품을 이용하여 결과를 비교하였다. 이 연구 결과를 통해, 균일하지 않은 read depth가 HLA typing에 부정적인 영향이 있음을 확인할 수 있었다.
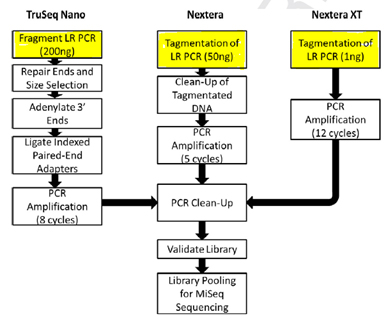
그림 1. Comparison of three Illumina library construction protocols. Covaris M220 was used to fragment DNA in TruSeq Nano.
2) Mayor, Neema P., et al. "HLA typing for the next generation." PLoS One 10.5 (2015): e0127153. 내 발췌
대립 유전자 수준에서의 HLA typing을 한번에 진행하는 것은 조직 호환성을 확인하는 연구에서 매우 이상적이다. 대량으로 진행되는 대부분의 molecular HLA typing 접근법의 결과로 분석하기 어려운 DNA 서열의 다형성 검출을 어렵게 한다. 보다 정확한 방법을 이용하는 경우에는 종종 시간과 비용에 한계가 있다 Pacific Bioscience는 Single Molecule Real Time (SMRT) DNA sequencing를 이용하여 대량으로 보다 정확한 HLA typing을 할수 있음을 확인하였다. SMRT sequencing 방법은 3 kb 이상의 HLA class Ⅰ 유전자 서열을 Takara LA Taq® 을 이용해 long-range PCR한 후, 긴 길이의 reads를 예외 없이 분석함으로써 다형성을 띄는 서열을 확인할 수 있다.
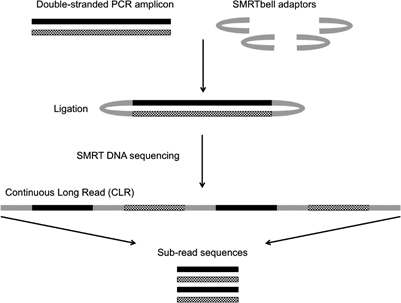
그림 1. Basic stages of the Single Molecule Real-Time (SMRT) DNA sequencing method.
PrimeSTAR® GXL DNA Polymerase
1) Liu, Chang, et al. "Accurate typing of human leukocyte antigen class I genes by Oxford Nanopore sequencing." The Journal of Molecular Diagnostics 20.4 (2018): 428-435. 내 발췌
HLA typing은 장기 기증자와 수혜자 간의 면역 반응 확인을 위해 매우 중요하다. 물론 빠르고 정확한 HLA typing 방법을 이용하는 것이 장기 기증을 위해 가장 이상적이나, Sanger sequencing이나 NGS을 이용하는 것은 기술적인 한계가 있었다. 대부분의 Illumina 혹은 ion-torrent의 HLA typing 방법은 long-range PCR product를 enzyme 처리한 후 증폭을 한번 더 진행하여, 총 분석 시간이 길 뿐만 아니라 GC 서열을 다수 포함하는 exon 부위에서 편향성이 확인될 수 있다. 본 논문은 인간 DNA 샘플로부터 정확한 class Ⅰ HLA 유전자를 분석하기 위해서는 Oxford nanopore sequencing이 최적화되었다는 것을 증명하고자 하였다. 이를 위해, PrimeSTAR® GXL DNA polymerase를 이용해 먼저 Class Ⅰ HLA 유전자 중 HLA-A, -B, -C 서열을 long-range PCR로 증폭하였으며, sequencing adaptor를 ligation한 뒤 MinION sequencing을 진행하였다.
2) Ozaki, Yuki, et al. "Cost-efficient multiplex PCR for routine genotyping of up to nine classical HLA loci in a single analytical run of multiple samples by next generation sequencing." BMC genomics 16.1 (2015): 318. 내 발췌
NGS를 이용한 HLA genotyping은 PCR, NGS, allele assignment 총 세 과정으로 진행된다. PCR-sequence specific oligonucleotide primers (SSOP)나 sequence based typing (SBT) 방법과 비교했을 때, PCR-NGS는 매우 노동 집약적이고, 시간이 많이 소요된다. 여러 DNA 샘플에서의 NGS 기반 HLA genotyping 방법을 간소화하고 가속화 하기 위해서, 본 논문의 저자들은 먼저, 다형성의 exon을 genotyping할 수 있는 HLA loci 타겟의 middle ranged PCR primer set를 새로 디자인하였고 PrimeSTAR® GXL DNA polymerase를 이용해 multiplex로 PCR 증폭하였다. 이를 이용해 HLA-A, HLA-B, HLA-C, HLA-DRB1/3/4/5, HLA-DQB1, HLA-DPB1을 포함하는 총 9개의 HLA loci를 대상으로 genotyping을 위한 multiplex PCR 방법을 개발하고 평가하였다.
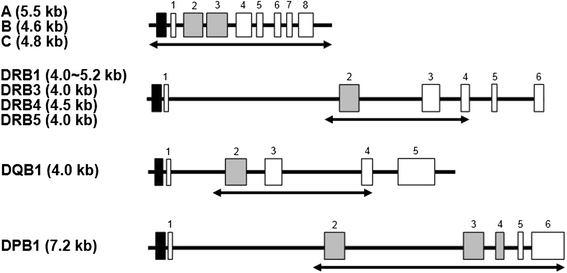
그림 5. Outline of the targeted PCR regions in nine HLA loci. Black, gray and white boxes indicate promoter regions, highly polymorphic exons and the other exons, respectively.
3) Yin, Yuxin, et al. "Application of high-throughput next-generation sequencing for HLA typing on buccal extracted DNA: results from over 10,000 donor recruitment samples." PLoS One 11.10 (2016): e0165810.내 발췌
Hematopoietic stem cell transplantation (HSCT)에서의 정확한 HLA typing을 하는 것은 HLA 질병 및 장기 이식 연구에서 매우 중요하다. 하지만, 최근의 Molecular typing 방법은 HLA 유전자의 항원 인식 부위 (antigen recognition site, ARS) 만을 확인하기 때문에, 결과적으로 많은 cis-trans 형태가 불분명하여 추가적인 typing 방법으로 이를 확인해야만 했다. 본 논문의 저자들은 National Marrow Donar Program (NMDP) 골수 기증자 10,063명을 대상으로 HLA typing을 진행하였다. 먼저 획득한 Swap DNA를 정제한 후, PrimeSTAR® GXL DNA polymerase와 HLA class Ⅰ 유전자 전체 서열을 타겟하는 PCR primer로 long-range PCR을 진행 후, NGS로 이를 분석하여 결과를 도출하였다.
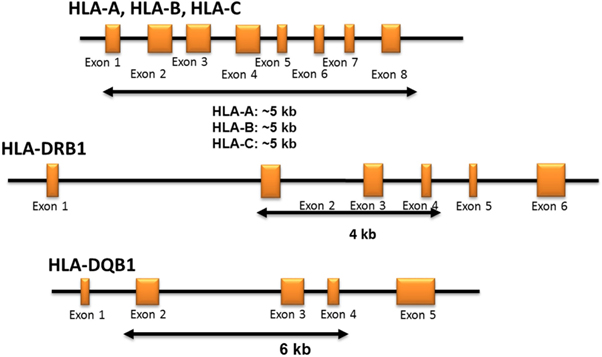
그림 1. HLA typing strategy.
The primer design captures full-length HLA class I genes (HLA-A, -B, -C) and exons 2-4 of DRB1 and DQB1 genes. There are two multiplex primer sets: Class I primer mix includes HLA-A, -B, -C, and Class II primer mix includes DRB1 and DQB1.