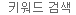- 고객지원
- 업무안내│제품문의
- 전화번호│02-2081-2510
- Email │support@takara.co.kr
- 업무시간안내
- [ 평 일 ] 09 : 00 ~ 18 : 00 │ [ 점심시간 ] 12 : 00 ~ 13 : 00
- 토·일요일, 공휴일은 휴무입니다.
경제적이고 효과적인 targeted NGS를 위한 긴 단편 증폭

경제적이고 효과적인 targeted Next-gen sequencing을 위한 긴 단편 증폭
(Long PCR for cost-effective and efficient targeted next-gen sequencing.)
(Long PCR for cost-effective and efficient targeted next-gen sequencing.)
자료 제공
Laboratory of Dr. Kai Wang, University of Southern California
Published paper
Jia, H., Guo, Y., Zhao, W. & Wang, K. Long-range PCR in next-generation sequencing: comparison of six enzymes and evaluation on the MiSeq sequencer. Sci. Rep. 4, 5737 (2014).
■ Introduction
Targeted sequencing: 큰 연구실에만 필요한 것이 아닌 보편적이고 강력한 기술
(a powerful technique that's not just for big labs)
Targeted sequencing (targeted resequencing이라고도 함)은 차세대 시퀀싱 (NGS)의 강력한 기능을 사용하여 게놈의 한정된 관심 영역을 조사하는 것이다. 이는 게놈 DNA의 특정 부분을 분석하고자 하는 중개의학 연구자에게 유용한 접근 방식이다. 임상 연구에서 게놈의 일부 작은 영역이 질병의 발생 또는 진행에 원인이 되는 것으로 알려진 경우, 이 영역의 서열 분석은 매우 유용하다. 또한 여러 대상자에서 관심 영역의 ultra-deep sequencing을 통해 이전에 알려지지 않은 희귀 변종 (rare variants)을 발견할 수도 있다. MiSeq (Illumina)과 같은 "개인용 (personal)" 게놈 시퀀서의 도입으로 중소 규모의 실험실에서 직접 NGS를 사용할 수 있게 되었으며, 이러한 연구 그룹은 최대치 유연성과 빠른 처리 시간이 필요한 조사의 최첨단(leading edge)에 위치할 수있다. 이 경우, 반응 최적화의 필요성을 줄이는 것은 가설을 신속하게 평가하는 데 매우 중요한 요소가 될 것이다.
■ Results
Proof of concept: polymerase 선택의 중요성(polymerase choice matters)
본 논문의 저자는 본 실험에서 4개의 회사에서 판매하는 6개의 PCR polymerase를 이용하였다. 각 polymerase는 각 제조사에서 권장하는 표준 프로토콜에 따라 BRCA1 (3개 단편 - 12.9 kb, 9.7 kb, 5.8 kb) 증폭에 사용되었다 (모든 효소에 대해 동일한 프라이머 세트사용, Amplicon Tm ; 54°C, 63.3°C, 54.5°C). 사용한 모든 효소 중에서 PrimeSTAR® GXL DNA Polymerase만이 3개의 단편을 모두 증폭하였으며 (그림 1, Panel D), 시간 절약이 가능한 2 step PCR protocol을 적용할 수 있는 유일한 효소였다.
그림 1. 6개의 효소를 이용하여 목적 유전자인 긴 단편을 증폭한 전기영동 결과
목적유전자의 사이즈는 아래와 같다.
- 12.9 kb (amplicon 1.1), 9.7 kb (amplicon 1.6), 5.8 kb (amplicon 2.8)
Panel A & Panel B. Polymerase 1 : 각각의 specific한 annealing temperature와 extension 시간을 적용하여 3개의 amplicon 확인 (상세 프로토콜 - Materials and Methods of Jia et al. 2014).
Panel C. Polymerase 2 : Brca1.1 과 2.8 이 유사한 Tm 값에서 증폭 확인
Panel D. PrimeSTAR GXL DNA Polymerase : 3개의 amplicon이 하나의 2 step PCR protocol에서 증폭.
Panel E. Polymerase 4 : Brca1.1과 2.8 증폭
Panel F. Polymerase 5, Polymerase 6 : Brca2.8 만 증폭
[Figure re-published from Jia et al. 2014 under Creative Commons license 4.0.]
The value of drama-free PCR amplification
증폭이 필요한 목적 유전자가 여러 개일 경우, PCR 조건의 최적화 과정은 적을수록 좋다. 이와 관련하여 이 논문의 저자들은 이번 테스트에 사용한 효소들 사이에서 심오한 차이를 발견하였다. 대부분의 효소는 여러 개의 목적 유전자 증폭시 최적화가 필요하였다. 하나는 각각의 target에 대해 서로 다른 cycling condition이 필요했고, 다른 하나는 최적화를 수행한 후에도 증폭이 되지 않았다. 그러나 PrimeSTAR® GXL DNA polymerase는 이러한 번거로움이 없었다. " PrimeSTAR® GXL DNA polymerase는 3 가지 target을 모두 증폭하기 위해 하나의 2 step PCR protocol을 사용할 수 있으며, 하나의 PCR기기를 사용하여 모든 target을 동시에 증폭할 수 있으므로 실제 설정에서 PCR을 위한 실험 설계 및 구현이 훨씬 쉬워진다." 라고 본 논문의 저자는 언급하였다.
3 step PCR protocol이 아닌 2 step 조건을 사용하는 것도 PCR 기기 사용시간을 줄일 수 있는 장점이다. 저자는 이 점을 강조하면서 "우리 실험에서 Takara PrimeSTAR® GXL DNA Polymerase를 사용하면 실험 조건을 변경하지 않고도 BRCA1/2의 모든 유전자를 증폭할 수 있다는 것을 발견하였다. 이것은 PCR 기기와 같이 resource가 한정된 상황에서 이 효소를 사용할 때 큰 장점이다."라고 언급하였다.
Improving targeted sequencing workflows: sequencing BRCA1 and BRCA2
조건 검토 실험이 매우 고무적이었지만, 연구자들은 보다 심층적인 연구에서 이 long-range PCR이 적합한지 확인하고자 하였다. 이를 위해서 피험자 9명의 DNA를 준비하여 BRCA1 과 BRCA2 의 전체 게놈 영역을 증폭하였다. 대상자 중 8명은 대조군 (유방암 없음)이었고, 9번째 환자는 유전성 유방암을 가지고 있었다. 이 실험의 목적은 실험 조건이 샘플의 그룹사이에서 일관되게 적용되는지와 양성 변이가 안정적으로 구분되는지를 평가하는 것이었다.
PrimeSTAR® GXL polymerase을 이용한 long-range PCR은 매우 성공적이었다 (그림 2). 모든 샘플에서 BRCA1/2 amplicon을 성공적으로 증폭할 수 있었으며, non-specific band나 끌림 현상없이 목표로 하는 단일밴드가 증폭되었음을 확인하였다.
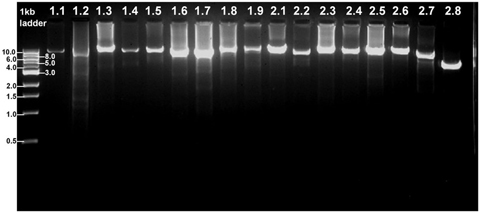
그림2. BRCA1 과BRCA2의 targeted sequencing analysis를 위하여 PrimeSTAR® GXL DNA Polymerase를 이용한 Long-range PCR 수행 결과. Amplicons 1.1–1.9은 BRCA1, amplicons 2.1–2.8 은 BRCA2 증폭.
Figure republished from Jia et al. 2014 under Creative Commons license 4.0.
Amplicon을 정제하여 sequencing을 위한 library를 준비하고 정량하였다. 9개의 normalized library를 pooling하여 Illumina MiSeq platform으로 sequencing하였다. 그 결과 평균 커버리지는2261X (range: 1285X ~ 3583X)이고, target region의 93.75 % (range: 81.55% ~ 100.00%)가 10 이상의 커버리지를 보였으며, target region의 98% (range: 92.53% ~ 100%)가 1회 이상 커버되었다. 샘플당 평균 234개의 SNV가 확인되었고, 대부분 non-coding variant였다. 일반적으로 알려진 질병관련 변이 (c.5946delT in BRCA2)는 유전성 유방암을 가지고 있는 환자에게서만 검출되었다. 흥미롭게도 대조군 샘플에서 의미를 파악하기 어려운 non-frameshift deletion이 확인되었다.
The use of long-range PCR in targeted sequencing
시퀀싱 분석과 결합하면, long-range PCR은 더 높은 감도를 구현할 수 있으며 유전자 변이를 검출하기 위한 더 빠르고 경제적인 도구가 될 수 있다. Long-range PCR에는 기존 효소보다 더 큰 처리 능력을 가진 중합효소가 필요하며, 이러한 기술의 발전 (polymerase 측면에서)은 genome mapping과 시퀀싱을 위한 PCR의 속도개선 및 간소화를 가져왔고, 분자유전학 연구를 촉진하였다.
물론 custom capture arrays와 같이 targeted sequencing을 위한 PCR을 사용하지 않는 (PCR-free) 대안도 있다. Jiaet al.은 2개의 다른 시스템을 사용하여 BRCA1/2용 캡처 솔루션을 설계하여, 이 두 개의 custom capture arrays의 성능을 서로 비교하고, long range PCR과도 비교하였다. 그들은 샘플 전체에 걸쳐 불균일한 시퀀싱 뎁스(uneven sequencing depth)가 있는 multiplex sequencing의 경우에도 최대 100.00%의 커버리지를 얻을 수 있음을 보여준 long-range PCR에 비해 custom capture system은 intron 및 exon 영역의 커버리지가 감소했음을 발견했을 뿐만 아니라, custom capture systems이 PrimeSTAR® GXL polymerase을 사용한 long-range PCR보다 비용이 4배 더 높다는 것을 확인하였다.
■ Conclusions
Concluding thoughts: a practical approach to targeted sequencing
Long-range PCR은 targeted sequencing에 중요한 역할을 한다. 그 이유는 분석이 필요한 샘플 수가 적고 (인트론을 포함한 전체 유전자 영역과 같은) 동일한 영역에 대한 분석이 필요할 때, 공급업체에 별도의 설계를 맡길 필요가 없이 소규모 실험실에서도 적용할 수 있기 때문이다. 본 저자들의 연구 목적은 중개의학연구를 포함한 다양한 응용 연구 분야에 이 방법을 적용할 수 있도록 실용적인 정보를 제공하는 것으로, intron을 포함한 전체 유전자 영역에 관심이 있을 때 NGS를 위한 long-range PCR을 사용하는 방법에 대해 실용적인 지침을 제공하고자 하였다.