Customer spotlight: profiling transcription factors with CUT&RUN sequencing
Date: January 11, 2019
Author: Takara Bio Blog Team
Categories: NGS
펜실베니아 대학의 Ph.D Karl Glastad가 NGS Library 제작을 위하여 다카라바이오의 ThruPLEX® Chemistry를 CUT&RUN 기법에 접목한 내용을 소개합니다.
ChIP-seq의 한계 극복
DNA 결합 단백질 (DNA-binding protein)은 세포 내에서 필수적이고 다양한 역할을 수행하고 있으며, 이에 대한 근본적인 메커니즘을 이해하고자 하는 연구자들에게 매우 중요한 실험 자원으로 여겨지고 있다.
일반적으로 DNA 결합 단백질의 상호작용을 분석하고 단백질 결합부위의 유전자를 규명하기 위하여 ChIP-seq을 이용한다. ChIP-seq은 차세대염기서열분석법 (NGS)과 크로마틴면역침강법
(chromatin immunoprecipitation; ChIP)을 결합한 방법이다. 하지만, ChIP-seq은 샘플양과 분석 타겟의 유형에 따라 한계점을 가지고 있다.
이에 반해, CUT&RUN sequencing (Cleavage Under Targets and Release Using Nuclease)은 ChIP-seq의 한계점을 극복하여 연구자들이 더 많은 수의 genome sequence를 더 쉽게 분석할 수 있도록
개선한 분석 기법이다. 다카라바이오는 펜실베니아 대학의
Shelley Berger 연구소에서 개미의 역할분담 (ant castes)의 후성유전학을
연구하는 박사 후 연구원, Karl Glastad와 함께 본 기술에 대하여 토론하였다.

Karl Glastad, PhD
현재 당신의 중점 연구는 무엇입니까?
우리는 개미의 역할분담 (카스트; caste)을 결정하는 분자에 대한 연구를 진행하고 있습니다. 구체적으로, 매우 밀접한 연관이 있는 각각의 개미가 매우 다른 환경적 자극 하에서 어떻게 완전히
다른 유형의 표현형을 발현하는 지에 대해 관심이 있습니다. 우리 연구의 주요 초점 중 하나는 ‘개미 서식지 내에서 역할분담 (카스트) 간의 극단적인 수명 차이 연구’ 입니다. 예를 들어,
하나의 개미 개체는 1년 이내로 생존하는 반면 그 개체의 누이는 10년 - 20년을 생존합니다. 이러한 개미 역할분담 간의 수명 차이의 주요 원인은 크로마틴 변이 (differential chromatin modification)와
전사인자의 결합 (transcription factor binding; TF binding)의 차이에서 기인합니다.
CUT&RUN 기법은 당신의 연구에 어떻게 적용됩니까? 본 기법이 기존의 기술 대비하여 좋은 점은 무엇입니까?
각 개미 개체 조직의 세포의 양 (15 - 150,000 cells)은 매우 적기 때문에 cross-linking ChIP-seq (X-ChIP)과 같은 기존의 방법으로 분석하는 것은 엄두를 내지 못 합니다. 반면, Native ChIP은 이러한 샘플에서
히스톤 변형을 분석하는 데 상당히 효과적이지만, 전사인자 (transcription factor; TF)와 같은 비히스톤 크로마틴 결합 단백질 (non-histone chromatin-binding proteins)을 분석할 수는 없습니다.
우리는 CUT&RUN 기법을 이용하여 기존의 X-ChIP으로는 분석이 불가능했던 전사인자 (TF)를 프로파일링 할 수 있게 되었습니다. 게다가, 개미에서 X-ChIP으로는 확인이 불가능했던 포유류 단백질의
보존영역 (conserved region)에 대해 생성된 몇 가지의 항체가 CUT&RUN에서는 적용이 가능함을 확인하였습니다. 따라서, 우리는 CUT&RUN 기법을 이용하여 개미 뇌의 여러 종류의 전사인자 (TF)를
확인할 수 있었을 뿐만 아니라, 포유류 항원/단백질에 대해 생성된 항체를 이용하여 대상 분자의 레퍼토리를 확대분석 할 수 있게 되었습니다.
CUT&RUN의 원리에 대해 설명해주시겠습니까?
기존의 ChIP 기법이 sonication 또는 핵산분해효소 활성 (nuclease activity)을 이용하여 게놈을 조각낸 후 목적단백질에 결합된 유전자좌위 (loci)를 침강시키는 것과는 달리, CUT&RUN 기법은
손상되지 않은 온전한 세포 (intact cell) 또는 핵 (nuclei) 내에서 항체를
in situ binding 시킨 후, 목적 단백질과 결합된 DNA를 절단합니다. CUT&RUN은 Protein A-MNase fusion protein을
이용하는데, 이는 목적 단백질과 결합된 DNA만 특이적으로 절단하므로 면역침강법 (immunopreciptation)과 같은 기존의 분석 방법에서 흔히 발생할 수 있는 background를 크게 감소시킬 수 있습니다.
또한, CUT&RUN은 면역형광법(IF)과 같이 손상되지 않은 세포 또는 핵에서 실험을 진행하기 때문에 기존의 ChIP에서 적용할 수 없었던 항체도 효과적으로 적용 가능합니다.
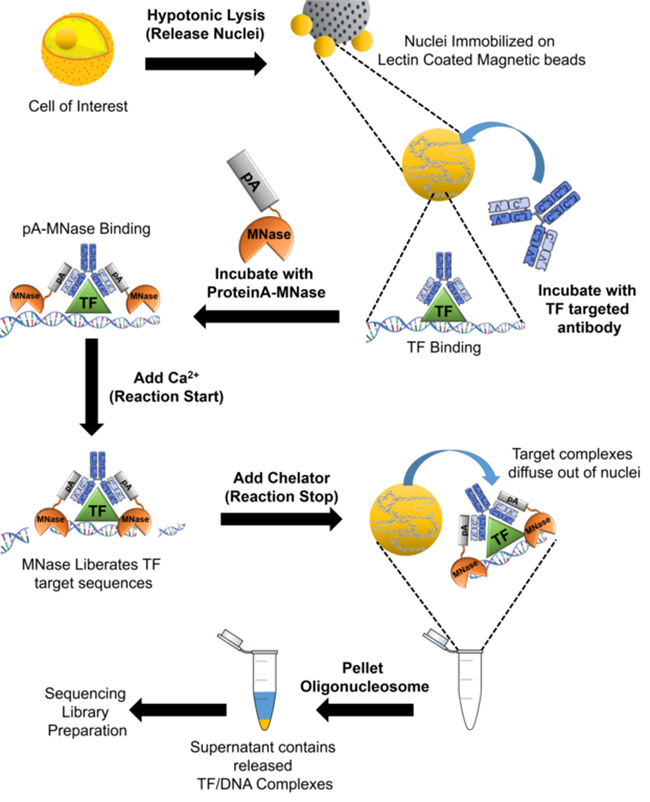 그림. CUT&RUN-sequencing workflow
그림. CUT&RUN-sequencing workflow (Figure by
Mannan369, used under CC BY-SA 4.0.)
CUT&RUN이 가장 적합하게 사용될 수 있는 분야는 무엇입니까? 때로, ChIP-seq과 같은 기존의 방식이 더 나은 경우도 있습니까?
CUT&RUN은 샘플의 양이 매우 적은 경우, 또는 타겟 항체가 면역형광법 (IF)에서는 적용이 가능하지만 면역침전 (IP)에서는 적용이 불가능한 경우에 적합합니다. 또한, CUT&RUN은 표적특이적으로 절단하기 때문에
기존의 ChIP-seq보다 background가 훨씬 낮습니다. 하지만, CUT&RUN기법은 아직 초기 개발 단계이므로 ChIP-seq이 제공하는 정보학적, 전산적인 자원의 이용이 어렵습니다.
예를 들어, CUT&RUN의 normalization control은 제한적이며, 서로 다른 조건 (특히, binding에 있어서 점진적인 차이가 있는 조건)을 비교하는 가장 좋은 방법에 대해서는 여전히 논의 중인 사안입니다.
하지만, 이러한 CUT&RUN의 한계점은 기술이 보급되면서 그리고, 정보학적, 전산학적인 자원이 증가함에 따라 해결될 수 있습니다.
CUT&RUN으로 분석한 데이터를 보완하기 위해 어떤 다른 실험기법을 이용하십니까?
우리는 RNA-seq, smRNA-seq, hiChIP-seq을 이용하여 looping의 변화를 확인하고, ATAC-seq을 이용해 접근성에 대한 근본적인 변화를 확인하고 있습니다.
연구의 궁극적인 목적은 무엇입니까?
개미를 이용하여 인간의 수명을 늘리는 것입니다

■
극소량 DNA-seq을 위한 ThruPLEX® 원리
■
ChIP-seq, CUT&RUN-seq 개요
■
ThruPLEX® DNA-Seq Kit (Code R400674)
- High performance from low inputs - 미량의 샘플 (50 pg ~ 50 ng)로부터 Illumina® library 제작
- Fast and simple workflow - 튜브 이동 없이 단일튜브 (single-tube or single-well)에서 세 번의 실험 과정 (3 step)으로 제작 완료 (library 제작 시간: 약 2시간)
- Compatible with major target enrichment platforms






